Páginas web fáciles con Jmol usando la función “Exportar a
página web”
Traducida y adaptada por Angel Herráez (diciembre 2015, para Jmol 14.4)
Guía original
de Jonathan
Gutow.
La función “Exportar a página web” incluida en Jmol produce de forma automatizada páginas web sencillas que muestran vistas que diseñas usando la aplicación Jmol. Puedes utilizar un editor de páginas web para cambiar el texto de las páginas así creadas, añadirles imágenes, tablas, etc. Tales páginas generadas automáticamente son asimismo un buen punto de partida para hacer páginas más complejas que requieren edición manual del código. Hay también soluciones alternativas para generar páginas web basadas en Jmol, que requieren software adicional o bien funcionan desde servidores. Actualmente la función “Exportar a página web” incluye dos plantillas sencillas:
“Pop-in”
Esta plantilla permite crear una página con varias imágenes de las vistas elegidas en Jmol. Cuando el usuario pulsa sobre una de esas imágenes, ésta se convierte en un objeto Jmol (de modo predeterminado, en su modalidad JSmol/HTML5) en la que el modelo se puede girar, ampliar, etc.
|
“ScriptButton”
Esta plantilla muestra un único objeto Jmol en la zona derecha de la ventana del navegador. A la izquierda queda una región, con barra de desplazamiento vertical, que contiene los botones mediante los cuales el usuario puede seleccionar las vistas.
|
Algunas mejoras que posiblemente se añadan en un futuro: 1) adición de botones opcionales para tareas comunes como girar la molécula o cambiar el color de fondo; 2) una nueva plantilla para una tabla de miniaplicaciones Jmol; 3) otras peticiones de los usuarios.
Para crear páginas con uno de estos formatos debes iniciar Jmol y abrir un archivo con la información molecular que quieres ver (Jmol lee muchos formatos de archivo, incluyendo .pdb, .cif, .mol y los formatos de salida de Gaussian y Gamess).
Para esta guía usaremos un archivo de salida en formato .log de Gamess, resultado de la optimización de geometría del etano en conformación alternada. Si quieres seguir el proceso con este ejemplo, descarga el archivo ethane-staggered-3-21G.log (para evitar que el navegador muestre el archivo, quizá necesites pulsar el botón derecho —o, en ratones con un solo botón, pulsar éste mientras mantienes pulsada la tecla Ctrl— y elegir “guardar como...”; también puedes copiar del navegador el contenido del archivo mostrado y pegarlo en un documento de texto).
Muchas de las páginas de esta guía se han generado usando la propia función de Jmol “Exportar a página web”.
Para comenzar (inicio de Jmol y apertura de un archivo):
Puedes pasar directamente a la sección “creación de una página web” si ya sabes cómo iniciar Jmol y abrir estructuras en él.
- Si no lo has hecho ya, descarga Jmol:
consigue la última versión estable en la sede web de Jmol (www.jmol.org). Una vez allí, sigue los enlaces de descarga. Cuando lo tengas, debes descomprimir el archivo descargado; necesitas extraer el archivo Jmol.jar y el archivo jsmol.zip. A continuación, descomprime este último.
- Asegúrate de que tienes instalado Java (una “Máquina Virtual de Java”, o JVM). Esta viene incluida por defecto en los sistemas con MacOS X. En la mayoría de sistemas Windows o Linux debes instalar la JVM tú mismo. Para ello, sigue las instrucciones de tu sistema en concreto. Se recomienda usar la versión Java ofrecida por Oracle
 .
.
-
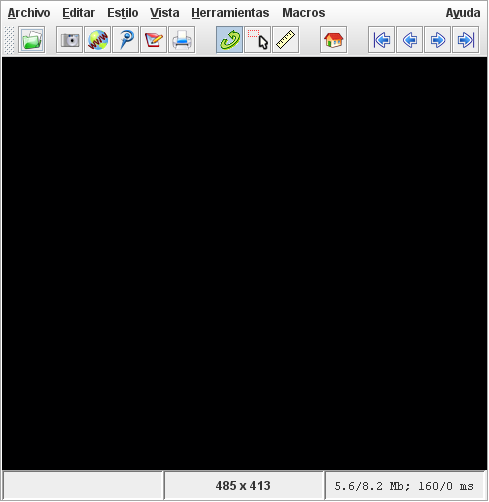
Inicio de Jmol. En tu ordenador, localiza el archivo Jmol.jar y ejecútalo. Una vez que se inicie el programa, tendrás una ventana con el aspecto de la figura 3.
- En los sistemas con MacOS X,
basta con hacer doble clic en el archivo para iniciar el programa.
- En los sistemas con Linux, al hacer doble clic en el archivo posiblemente se intente descomprimir el archivo jar. Si fuera ese el caso, necesitarás iniciarlo desde un terminal o “línea de comandos” (escribiendo
java -jar Jmol.jar) o bien usar la función “Abrir con...” de la interfaz gráfica del sistema, que generalmente está disponible haciendo clic sobre el icono con el botón derecho del ratón.
- En Windows puede ser difícil identificar el archivo, pues por defecto las extensiones están ocultas y hay varios archivos con nombre Jmol y con extensiones diferentes. Para mostrar las extensiones, ve al menú “Herramientas” y elige “Opciones de carpeta...”, selecciona la pestaña “Ver” y baja hasta encontrar la opción “Ocultar las extensiones de archivo para tipos de archivo conocidos”. Quita la marca de esta opción y pulsa “Aceptar”.
Intenta ejecutarlo haciendo doble clic sobre el icono de Jmol.jar; en caso de que esto no funcione (depende de la versión de Windows), quizás puedas conseguirlo usando javalauncher .
Si no es así, pulsa con el botón derecho sobre el icono, elige “Abrir como...” y selecciona “Java Platform”. Finalmente, si esto tampoco funcionase a pesar de que tienes instalada una máquina virtual de Java, abre una ventana con el “símbolo del sistema” o “línea de comandos” (
.
Si no es así, pulsa con el botón derecho sobre el icono, elige “Abrir como...” y selecciona “Java Platform”. Finalmente, si esto tampoco funcionase a pesar de que tienes instalada una máquina virtual de Java, abre una ventana con el “símbolo del sistema” o “línea de comandos” (C:\>), cambia a la carpeta adecuada y escribe java -jar Jmol.jar.
- Apertura de archivos. En el menú “Archivo” (barra superior de la ventana de Jmol) elige “Abrir...”. Utiliza el diálogo para desplazarte a la carpeta en la que esté el archivo deseado, selecciona éste y pulsa en “Abrir”. Si el archivo es de un tipo reconocido por Jmol, se abrirá y aparecerá un modelo de la molécula.
Creación de una página web
- Familiarízate con Jmol.
Si ya sabes cómo hacer que Jmol muestre mediciones y orbitales,
cómo cambiar el tamaño de los átomos y cosas así, pasa a la etapa 2. Si no es así,
visita estos enlaces para aprender cómo hacer que Jmol muestre la molécula del modo que te interesa:
( indica un enlace a un sitio web externo, que se abrirá en una ventana nueva)
indica un enlace a un sitio web externo, que se abrirá en una ventana nueva)
Tamaño
| Rotación
| Giro continuo
| Tamaño de los átomos
| Grosor de los enlaces
| Mediciones
| Representaciones esquemáticas de biomoléculas
| Mostrar orbitales
| Instrucciones en la ventana de guiones
| Navegación por un archivo multi-modelo
| Adición de texto
| LCAO esquemáticos | Mostrar información cristalográfica
| Mostrar información cristalográfica | Documentación de guiones de Bob
Hanson
| Documentación de guiones de Bob
Hanson | Mucho más sobre Jmol y sus prestaciones (en la Wiki de Jmol)
| Mucho más sobre Jmol y sus prestaciones (en la Wiki de Jmol) .
.
- Creación de páginas web utilizando el diálogo “Generador de páginas web de Jmol”.
- Procedimiento general, común a todas las plantillas.
- Abre el diálogo del “Generador de páginas web de Jmol” pulsando en el icono
 “Exportar a página web...” o bien seleccionando el menú “Archivo” > “Exportar” > “Exportar a página web”. Aparecerá un cuadro de diálogo.
“Exportar a página web...” o bien seleccionando el menú “Archivo” > “Exportar” > “Exportar a página web”. Aparecerá un cuadro de diálogo.
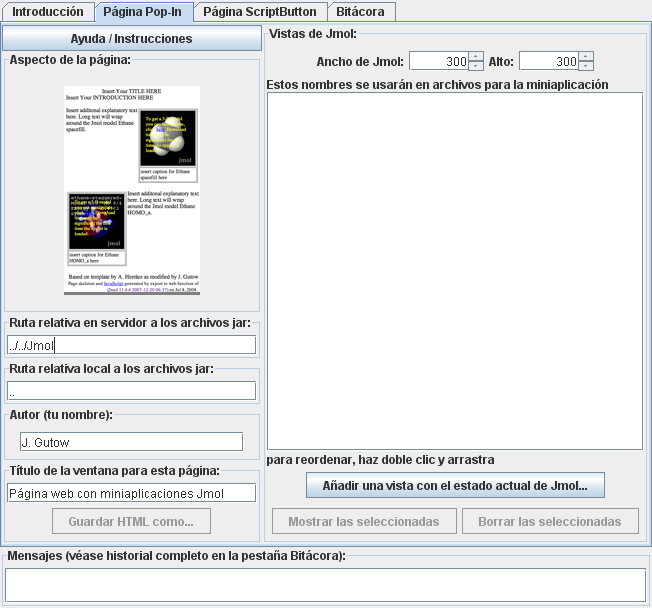
- Elige una de las plantillas pulsando en la pestaña respectiva (usaremos aquí la plantilla “Pop-in” como ejemplo). Consulta la figura 4.
- La zona izquierda del diálogo contiene información general necesaria para que los objetos Jmol funcionen en tu servidor (instrucciones para instalar Jmol en tu servidor). El botón “Ayuda/Instrucciones” proporciona una versión breve de la información de esta guía.
- En la primera caja de texto debes indicar la ruta (indicaciones para el directorio o carpeta) donde estén los archivos de Jmol en el servidor, relativa a la carpeta o directorio donde vaya a estar tu página web. Escribe un “../” por cada nivel que se deba ascender en la estructura de carpetas desde la ubicación de la página web, y a continuación escribe los nombres de las carpetas por las que haya que descender.
Ejemplo: supongamos que
JSmol.min.js esté en la carpeta “/jsmol” del servidor y vas a crear la carpeta “/estructuras/mipagina” para que contenga los archivos .html y otros archivos de datos.
La ruta correcta para los archivos de Jmol es entonces “../../jsmol” (dos niveles arriba y luego uno abajo hasta la carpeta jsmol).
- En la segunda caja de texto
debes poner la ruta relativa para los archivos de Jmol en tu ordenador. De este modo puedes probar las páginas sin necesidad de subirlas al servidor.
Esta es una configuración sencilla que recomendamos (es posible usar otras ubicaciones):
- Pon “../jsmol” en ambas rutas (local y servidor) y coloca tus páginas en carpetas "hermanas" de la carpeta jsmol.
- Pon tu nombre como autor.
- Escribe un título descriptivo para la página.
- Procedimiento específico para la plantilla “Pop-in”.
- Procedimiento específico para la plantilla “ScriptButton”.
Última actualización: 8 dic. 2015
por A. Herráez