15.6 Preparación de proteínas de fusión
El término proteínas de fusión —sería más correcto llamarlas proteínas fusionadas— se aplica a proteínas obtenidas mediante tecnología de DNA recombinante, de modo que una misma molécula polipeptídica combina regiones correspondientes a dos proteínas diferentes. Generalmente se puede entender como sinónimo el nombre proteínas quiméricas.
Las aplicaciones de estas proteínas son múltiples y dependen de las propiedades de las proteínas que combinan. Es difícil establecer una generalización, por lo que se describirán varios ejemplos ilustrativos.
El principio metodológico esencial es la formación de un DNA recombinante que encadene regiones de dos genes diferentes. Dicho recombinante se somete a clonación celular con expresión, lo que permite obtener la proteína.
Pulse sobre los títulos de apartado (fondo azul) para desplegar o replegar su contenido.
Proteínas etiquetadas
Se conoce como etiqueta una molécula, unida de modo covalente a la proteína, que ejerce de marcador reconocible.
Las etiquetas tienen dos aplicaciones:
- si la etiqueta es detectable sirve como marcador para localizar o incluso cuantificar la proteína (por ejemplo, para estudiar su ubicación subcelular, su transporte, la magnitud de su expresión...);
- si la etiqueta tiene la capacidad de unirse de forma específica a una segunda proteína (anticuerpo o proteína ligante), o bien a un ligando, permite la purificación selectiva de la proteína etiquetada de interés, empleando métodos de afinidad.
Existen etiquetas como biotina o digoxigenina (pág. 187-188) que, no siendo proteicas, no corresponden a la categoría de proteínas fusionadas. Sin embargo, otras etiquetas son péptidos, proteínas o fragmentos de éstas, y se incorporan fusionadas como parte de la cadena polipeptídica. Entre las más utilizadas pueden destacarse las siguientes:
Oligohistidina o etiqueta His
- Se trata de un péptido formado por entre 5 y 9 residuos de histidina. Posee propiedades quelantes, uniendo iones Ni2+ o Co2+ con elevada afinidad (µM). Además, resulta conveniente por su pequeño tamaño y por ser poco inmunogénico. Una vez cumplida su función puede retirarse de la proteína empleando ciertas endo- o exopeptidasas específiicas.
- Se incorpora a la cadena de la proteína; comúnmente se hace sobre el extremo N-terminal o el C-terminal, siempre que esté accesible (no orientado al interior de la proteína globular) y que su sustitución no perjudique la funcionalidad.
a) mediante clonación celular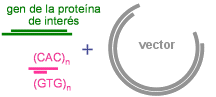 preparar DNA recombinante
preparar DNA recombinante
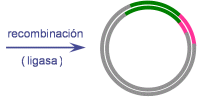
Como ejemplo, el oligonucleótido CACCACCACCACCACCAC codifica His-His-His-His-His-His
b) mediante PCR amplificar mediante PCR
amplificar mediante PCR
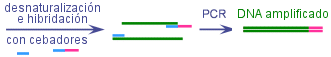
Uno de los cebadores incluye, por ejemplo, el oligonucleótido CATCATCATCATCATCATTAG que codifica His-His-His-His-His-His (C-terminal) - Permite la purificación mediante afinidad, sobre un soporte que posea quelantes de níquel o cobalto. Los más habituales son
el NTA y
el IDA, inmovilizados
por unión covalente a un soporte sólido (como partículas de agarosa o de un polímero sintético). La técnica se llama cromatografía de afinidad sobre metal inmovilizado
(IMAC).
Mostrar detalles
Posando el puntero sobre las distintas partes del dibujo se muestran más detalles.
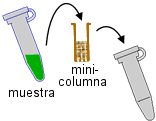
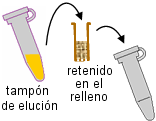
aplicar la muestra
a la columna
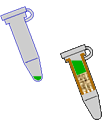


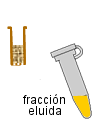
resultado 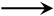
continuar 
La elución de la proteína unida al soporte se puede realizar empleando una concentración suficiente de imidazol (que compite con la histidina) o disminuyendo el pH para protonar los residuos de histidina (pH<6 para el caso del cobalto, pH<4 para el níquel). También puede emplearse un quelante fuerte como el EDTA que secuestre los iones Ni2+ o Co2+ del soporte.
Mostrar estructura 3D del quelato.
Octapéptido o etiqueta flag
- La secuencia DYKDDDDK, llamada etiqueta flag®, se añade a la proteína de interés empleando el mismo tipo de estrategia que para la etiqueta His. Se detecta o se captura gracias a anticuerpos específicos para el octapéptido. La enteroquinasa o enteropeptidasa (la misma proteasa responsable de la activación proteolítica del tripsinógeno) permite escindir la etiqueta una vez que la proteína se ha purificado.
GFP (proteína fluorescente verde)
- Se trata de una proteína de 27 kDa (268 residuos aminoácidos) que posee fluorescencia de modo natural. Se detecta habitualmente mediante microscopía y permite principalmente cuantificar la expresión o bien ubicar la localización de la proteína de interés. Además, existen actualmente formas recombinantes de la proteína (originaria de medusas) que emiten la fluorescencia de diferentes colores (GFP, BFP, CFP, YFP).
GST (glutatión-S-transferasa)
- Enzima proteica, globular, de 26 kDa (220 residuos aminoácidos). Cataliza reacciones entre el glutatión (tripéptido ECG) y diversos sustratos con centros electrófilos.
- Se incorpora su secuencia génica al DNA recombinante delante –o detrás– del gen de la proteína de interés, lo que conducirá a una proteína fusionada. En ocasiones se introduce un péptido LVPRGS intermedio, diana de la proteasa trombina, con el fin de poder cortar una vez purificada y recuperar así la proteína de interés sin etiqueta.
- Se puede detectar la proteína fusionada gracias a la actividad enzimática de la GST. Para la purificación de la proteína etiquetada se aprovecha la afinidad por el sustrato (glutatión); el grupo tiol del glutatión hace sencilla su fijación al soporte.
El procedimiento es análogo al mostrado más arriba para la etiqueta de oligohistidina: un soporte con glutatión unido de forma covalente, fijación selectiva de la proteína con etiqueta GST, elución mediante la adición, en este caso, de un exceso de glutatión libre.
Otras etiquetas proteicas
- Proteína ligante de maltosa (MBP, originaria de Escherichia coli). Permite la purificación sobre un soporte con amilosa. Se eluye con maltosa libre.
- Dominio ligante de quitina (CBD, 52 residuos aminoácidos, parte de la quitinasa de Bacillus circulans). Permite la purificación sobre un soporte de quitina. Se eluye con detergente o en medio alcalino.
- Dominio N-terminal (24 kDa, 220 residuos aminoácidos) de la subunidad B de la DNA girasa. Se puede purificar empleando novobiocina unida a un soporte de agarosa.
Proteínas bifuncionales
En ocasiones interesa combinar en una sola molécula las propiedades de dos proteínas.
Dominios de dimerización
Ejemplo 1:
Puede considerarse el mismo utilizado para explicar la clonación con expresión: la fusión de la proteína de interés con un dominio de la subunidad B de la DNA girasa, como forma de provocar la dimerización de aquélla. En el caso mostrado (el represor λ), la consecuencia es la unión al DNA y su actuación como factor de transcripción.
Ejemplo 2:
La quinasa de proteínas PKR, cuando se activa, fosforila la subunidad α del factor eIF-2, que es el factor eucariótico de iniciación de la traducción responsable de portar el Met-tRNAi hacia el ribosoma (pág. 332). Dicha fosforilación estabiliza el complejo de eIF-2 con eIF-2B impidiendo el intercambio de GDP por GTP (el reciclado de eIF-2, pág. 332 y web 21.5). Como consecuencia, no hay eIF-2:GTP disponible y se detiene la síntesis de proteínas. La función natural de este mecanismo regulador de la traducción consiste en que la presencia de RNA bicatenario (procedente de una infección vírica) desencadena la unión a él de la PKR en forma de dímero. Esta dimerización conduce a la autofosforilación de PKR, que se activa como quinasa, fosforila a eIF-2 y detiene la síntesis de proteínas. Se evita así la proliferación del virus.
Considerando este mecanismo, se ha preparado una proteína fusionando dominios de la PKR y la DNA girasa, para controlar experimentalmente la dimerización y así estudiar el proceso de activación de la PKR:
| dominio C-terminal (quinasa) de la PKR dominio N-terminal de la subunidad B de la girasa |
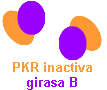
|
Anticuerpos quiméricos
El desarrollo de anticuerpos monoclonales se realiza predominantemente en líneas celulares de ratón. La uitlización experimental de dichos anticuerpos queda restringida, debido a la reacción inmunológica adversa, a los ratones. El interés de utilizar los anticuerpos monoclonales como agentes diagnósticos o terapéuticos en seres humanos puede cumplirse si se consigue combinar en una misma molécula
- la región que reconoce al antígeno propia de una IgG de ratón (el anticuerpo monoclonal de interés), con
- la región que es reconocida por receptores y por anticuerpos, propia de una IgG humana.
(La primera es una parte de la región variable, la segunda es principalmente la región constante.)
Dependiendo de qué parte de la molécula de inmunoglobulina fusionada proceda de cada especie, se los llama anticuerpos quiméricos o anticuerpos "humanizados":
|
Esquema de la estructura V = región variable L = cadena ligera (o liviana) |
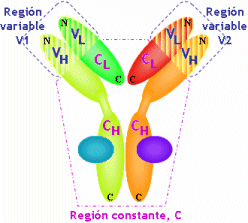

|
dominios de IgG de ratón |
La obtención del anticuerpo quimérico o el humanizado requiere, obviamente, la combinación de las regiones adecuadas de los genes de IgG de ambas especies y la clonación con expresión del DNA fusionado.
Proteínas simplificadas o modificadas
En ocasiones resulta conveniente preparar derivados de una proteína que combinan sólo algunas de sus partes, o que conectan en una sola cadena subunidades originalmente independientes, de modo que no puedan separarse. Se comentará un solo ejemplo:
Anticuerpos scFv
Se trata de un "fragmento variable monocatenario", o "fragmento Fv monocatenario". Resulta ventajoso en algunas aplicaciones experimentales porque mantiene la capacidad de unión al antígeno pero es una única cadena polipeptídica. Se prepara combinando las regiones génicas que codifican el domino variable de cada una de las cadenas de una inmunoglobulina, con un conector entre ambas que codifica un péptido corto flexible e hidrófilo (gracias a sus aminoácidos constituyentes, habitualmente Gly y Ser o Thr).
Ejemplo de preparación (puede verse más información posando el puntero sobre las diferentes partes del dibujo):
Este tipo de molécula, como ejemplo, constituye un fármaco llamado pexelizumab, que se emplea para reducir los efectos secundarios de la cirugía cardiaca. Se trata de un scFv preparado a partir de un anticuerpo humanizado dirigido contra la proteína C5, un componente del sistema del complemento, parte del sistema inmunitario.
Referencias: