12.5 Técnicas para analizar la interacción de ácidos nucleicos con proteínas
Se han ido desarrollando numerosos ensayos que guardan alguna similitud metodológica con la electroforesis, con las hibridaciones Southern (pág. 169) y northern (pág. 170) o con el inmunoensayo western (pág. 176). Se describen aquí algunos de uso más frecuente.
Ensayos de la interacción entre ácido nucleico y proteína
Ensayo southwestern
Se trata de encontrar proteínas ligantes de DNA que reconozcan en éste una determinada secuencia de nucleótidos.
- Se realiza una electroforesis de las proteínas, desnaturalizadas con SDS, en gel de poliacrilamida (SDS-PAGE). En esta técnica las proteínas, desplegadas, se separan de acuerdo con su número de residuos aminoácidos o –lo que es equivalente– con su masa molecular.
- Se realiza la transferencia a nitrocelulosa o material similar.
- Opcionalmente, se hace un bloqueo de la membrana con una proteína inerte, para reducir la adsorción de la sonda (ruido de fondo).
- Se añade una sonda bicatenaria de DNA, que posea la secuencia que se quiere ensayar. Para conseguir una mayor afinidad, puede prepararse un concatémero, es decir, una sonda donde la secuencia diana se repite varias veces consecutivas.
- Se incuba en condiciones adecuadas para la asociación (pH y fuerza iónica fisiológicos), se lava para retirar el exceso de sonda y se detecta la señal del marcaje de ésta.
Ensayo northwestern
Es un ensayo similar al anterior pero dirigido a encontrar proteínas ligantes de RNA.
Ensayo de retardo en gel
Se denomina también EMSA (de electrophoretic mobility shift assay, ensayo de cambio de la movilidad electroforética).
Permite determinar a qué moléculas o fragmentos de ácido nucleico se une una proteína de interés.
- Se realiza una electroforesis de los ácidos nucleicos, DNA o RNA. Se emplea un gel de agarosa o poliacrilamida dependiendo del tamaño de las moléculas o fragmentos a analizar (pág. 139). En una calle se coloca la muestra de ácido nucleico, en otra una mezcla de dicha muestra con la proteína a ensayar.
- Se revela el resultado, bien tiñendo el ácido nucleico con un compuesto adecuado (por ej., un intercalante) o bien detectando una etiqueta incorporada previamente al ácido nucleico.
Las moléculas de ácido nucleico que unan la proteína verán retrasado su avance por el gel (debido al mayor tamaño del complejo), con lo cual la banda correspondiente queda desplazada, más cerca del origen (menor avance, retardo).
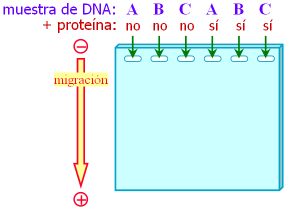 |
desarrollar la electroforesis |
Análisis e interpretación del resultado:
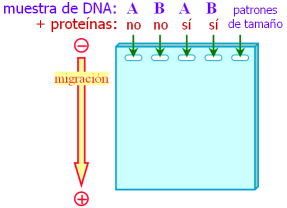
- La muestra A contiene un solo tipo de DNA (calle 1), que une la proteína estudiada (calle 4 distinta de la 1).
- La muestra B contiene un solo tipo de DNA (calle 2), que no une la proteína (calle 5 igual a la 2).
- La muestra C contiene varias moléculas de DNA distintas (calle 3); de ellas, la segunda más pequeña contiene la secuencia reconocida por la proteína (calle 6).
Huella con DNasa
Con esta técnica (en inglés, DNA footprinting) se determinan las posiciones de DNA a las que se unen una o varias proteínas. Por ejemplo, puede usarse para averiguar la ubicación de los promotores (pág. 284) a los que se unen factores de transcripción (pág. 287).
- Se prepara la muestra de DNA y se marca en uno de los extremos de una hebra. Puede hacerse esto mediante métodos de marcaje terminal por relleno o con polinucleótido quinasa. También puede usarse una sonda, si se conoce una secuencia próxima a uno de los extremos del DNA.
- Se incuba el DNA con las proteínas cuya capacidad ligante se desea ensayar. Por ejemplo, para estudios de promotores se puede añadir un extracto de proteínas nucleares (que contendrá todos los factores de transcripción).
- Se añade una nucleasa, típicamente la DNasa I en presencia de Mn2+, que corta los enlace fosfodiéster de ambas hebras. Aplicando condiciones limitantes (baja concentración de enzima, temperatura baja) cada molécula se corta en un número reducido de puntos, de forma aleatoria. La mezcla de fragmentos resultante cubrirá todo el intervalo de tamaños, salvo que las zonas donde se habían unido las proteínas quedan protegidas de la DNasa y no sufren cortes.
- Se realiza la electroforesis de los fragmentos de ácido nucleico.
- Se revela el resultado, gracias al marcaje incorporado inicialmente.
Como la marca está en un extremo, el tamño de los fragmentos es un indicador directo de la posición del corte (similarmente a lo que ocurre en la secuenciación del DNA). En consecuencia, el tamaño de los fragmentos ausentes indica las posiciones del DNA cubiertas por una proteína (la "huella" que ha dejado).
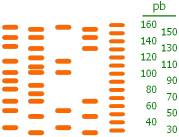
 |
desarrollar la electroforesis |
Análisis e interpretación del resultado:
- Las proteínas ensayadas se unen al DNA de la muestra A en una zona entre 50 y 100 nt del extremo de la molécula y en otra zona entre 120 y 160 nt.
- Y se unen al DNA de la muestra B en una zona entre 70 y 130 nt del extremo de la molécula.
Ensayos de la interacción entre varias proteínas
Ensayo far-western
El planteamiento es similar al ensayo western (pág. 176), pero la detección no se realiza con un anticuerpo, sino empleando otra proteína (denominada a veces el "cebo"). Pueden así localizarse las proteínas presentes en la muestra (las "presas") capaces de interaccionar con la proteína "cebo". La proteína "cebo" debe estar marcada para su detección, o bien disponerse de un ensayo para ella –quizás un anticuerpo–. Se puede considerar el "cebo" como un reactivo, o como una sonda aunque no en el sentido de hibridación.
Un caso frecuente es el empleo de una proteína de fusión, preparada mediante ingeniería genética combinando la proteína "cebo" cuya unión se quiere ensayar con una etiqueta que permita detectarla (pág. 186 ). Por ejemplo, se usan a menudo la etiqueta GST (glutatión-S-transferasa) y la etiqueta GFP (proteína fluorescente verde). La etiqueta puede tener una propiedad o actividad detectable, o estar marcada, o bien disponerse de un anticuerpo contra ella.
- Separación de las proteínas de la muestra mediante electroforesis SDS-PAGE (en gel de poliacrilamida con SDS).
- Transferencia a una membrana de nitrocelulosa o material similar.
- Bloqueo de la membrana con un exceso de proteína inerte (para evitar que la siguiente proteína se adsorba en las posiciones libres y eleve la señal de fondo).
- Incubación con la proteína de interés,
- marcada con un isótopo radiactivo,
- marcada con una etiqueta (como digoxigenina, biotina, oligohistidina, octapéptido flag) para la que se dispone de anticuerpo o proteína ligante específicos, o
- marcada con una enzima.
- Detección de la proteína de interés,
- mediante autorradiografía o una técnica homóloga,
- empleando una proteína ligante de la etiqueta (avidina o estreptavidina, anticuerpo), marcada a su vez, o
- mediante un ensayo enzimático con producto detectable (color, fluorescencia, quimioluminiscencia...).