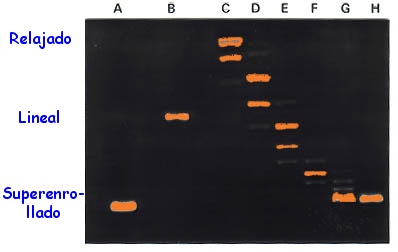
Plásmido![]() de 3000 pb, gel de agarosa al 1,75%, revelado con bromuro de etidio bajo
luz ultravioleta.
de 3000 pb, gel de agarosa al 1,75%, revelado con bromuro de etidio bajo
luz ultravioleta.
A= plásmido intacto (con su superenrollamiento natural).
B= plásmido abierto por corte con una endonucleasa.
C...H= plásmido tratado con concentraciones de topoisomerasa
y tiempos decrecientes.