La FISH es una tecnología que utiliza sondas de DNA marcadas con un fluoróforo para detectar o confirmar anomalías génicas o cromosómicas que generalmente están más allá de la capacidad de resolución de la citogenética de rutina. En primer lugar, la muestra de DNA (cromosomas metafásicos o núcleos en interfase) se desnaturaliza, proceso que separa las hebras complementarias de la estructura bicatenaria en doble hélice del DNA. A la muestra desnaturalizada se le añade entonces la sonda de interés, que se asociará al DNA de la muestra en el sitio diana, en el proceso denominado hibridación, donde se vuelve a formar una doble hélice. La sonda está unida covalentemente (marcada) con un fluoróforo, que emite una señal observable mediante un microscopio de fluorescencia; así la muestra de DNA se puede clasificar según la presencia o ausencia de la señal, lo que desvela la presencia o ausencia de la secuencia diana en el DNA cromosómico.
FISH de metafase
La FISH puede usarse sobre células en metafase para detectar microdeleciones específicas más allá de la capacidad de resolución de la citogenética de rutina, o para identificar material extra de origen desconocido. Puede también ser de ayuda en casos donde es difícil determinar mediante la citogenética de rutina si un cromosoma tiene una deleción simple o si está implicado en una reorganización sutil o compleja. Además, la FISH de metafase puede detectar algunos de los reordenamientos específicos que se observan en ciertos tipos de cáncer.
El número de síndromes de microdeleción diagnosticados por FISH está aumentando con rapidez. La sensibilidad de estos ensayos es en todos los casos mejor que la de la citogenética de rutina, pero depende del síndrome en concreto. En algunos, la sonda es específica para el gen defectuoso, como en el síndrome de Williams, donde el 96% de los pacientes con diagnóstico confirmado poseen una deleción en el gen de la elastina. En otros síndromes como el de Prader-Willi/Angelman, la etiología es heterogénea y las microdeleciones sólo existen en una parte de los casos (60%).
FISH de interfase
La FISH se puede usar sobre células en interfase para determinar el número de copias de uno o varios cromosomas, así como para detectar algunas reorganizaciones específicas que son características de ciertos tipos de cáncer. La ventaja principal de la FISH de interfase es que se puede realizar muy rápidamente si es preciso, normalmente en 24 horas, porque no requiere cultivo para la proliferación de las células.
Un buen ejemplo es el ensayo de aneuploidía, que se realiza sobre células del fluido amniótico cuando hay una fuerte indicación clínica de una de las trisomías más comunes. Se desnaturalizan los núcleos de la muestra, se hibridan con sondas específicas para los cromosomas 13, 18, 21, X e Y, y los resultados están disponibles habitualmente en 24 horas. La prueba de aneuploidía suele incluir también una citogenética de rutina para confirmar los resultados o detectar cualquier anomalía no detectada por la FISH de interfase.
Ejemplos de FISH
Pulse en los enlaces siguientes para ver algunos ejemplos de FISH detectados en el laboratorio de Dynacare:
Ejemplos de FISH de metafase
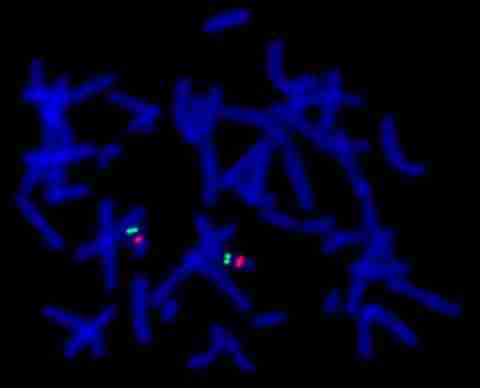
Éste es un ejemplo de una célula en metafase que se ha hibridado con la sonda diseñada para detectar el síndrome de DiGeorge, llamado también velocardiofacial (VCFS), CATCH 22 o de Shprintzen, causado por una microdeleción en el cromosoma 22. En este caso particular, se emplea una mezcla bicolor de dos sondas distintas para el cromosoma 22. La señal verde es un control interno que hibrida en 22q13. Permite identificar rápidamente los dos cromosomas 22. La señal roja está situada en la región DiGeorge, en 22q11.2. Puesto que ambos cromosomas 22 tienen la señal roja, en esta célula no hay microdeleción en la región DiGeorge y este paciente no padecería el síndrome de DiGeorge.
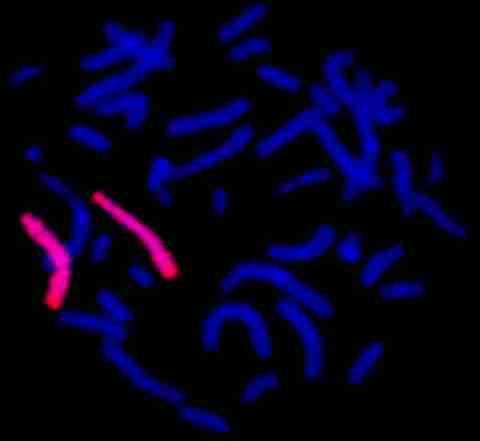
Esta otra metafase se ha hibridado con una sonda "de pintado" para el cromosoma 4 (una colección de sondas que se unen a lo largo de todo el cromosoma), lo que hace que el cromosoma completo emita fluorescencia. Uno de los cromosomas 4 de este paciente era anormal, pero era difícil determinar con la citogenética de rutina si tenía una pequeña deleción terminal en 4q o si era el resultado de una reordenación más compleja. Puesto que ambos cromosomas 4 muestran fluorescencia en toda su longitud y no hay material fluorescente en ningún otro cromosoma, esto sugiere que la anomalía es una deleción terminal pequeña. La metafase mostrada a continuación es del mismo paciente y confirma este diagnóstico.
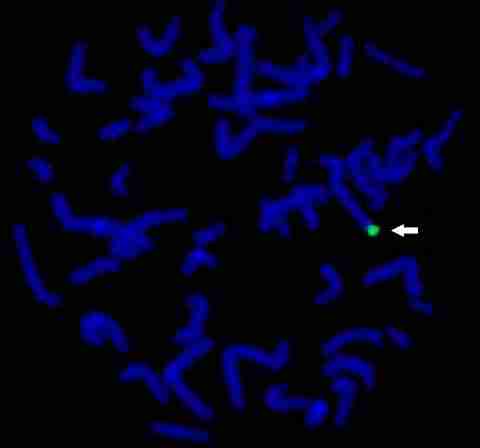
Esta metafase del mismo paciente anterior se ha hibridado con una sonda diseñada para la parte terminal del cromosoma 4q. Puesto que sólo hay una señal verde, se confirma que en uno de los cromosomas 4 falta material del extremo terminal del brazo q. Este caso es un buen ejemplo de cómo combinando la citogenética de rutina con la FISH se pueden diagnosticar con exactitud anomalías cromosómicas sutiles.
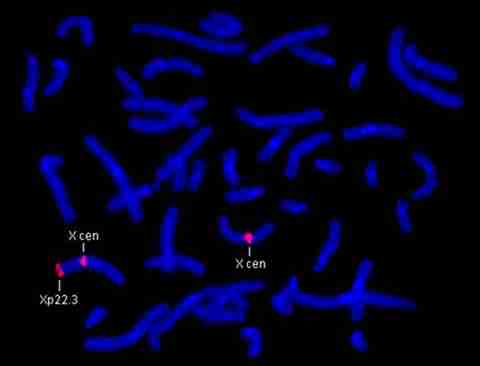
Éste es un ejemplo de una célula en metafase que se ha hibridado para detectar la deficiencia de esteroide-sulfatasa, causada por una microdeleción en el cromosoma X. En este caso particular, se emplea una mezcla de dos sondas independientes para el cromosoma X, ambas con color rojo. La señal de la sonda marcada con "X cen" es un control interno localizado en el centrómero de X, que permite la identificación rápida del (o los) cromosoma(s) X. La señal "Xp22.3" de la sonda está localizada en la región de la esteroide-sulfatasa, en Xp22.3. Puesto que hay dos cromosomas X y sólo uno tiene la señal del gen de la esteroide-sulfatasa, este individuo es una mujer portadora de la deficiencia genética de esteroide-sulfatasa.
Ejemplos de FISH de interfase
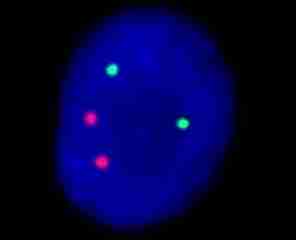

Éste es un ejemplo de la prueba de aneuploidía, donde a los núcleos en interfase procedentes de células de fluido amniótico se han añadido sondas de DNA diseñadas para los cromosomas 13, 18, 21, X e Y. El núcleo de la izquierda se ha hibridado con sondas para los cromosomas 13 (verde) y 21 (rojo). El núcleo de la derecha se ha hibridado con sondas para los cromosomas 18 (azul claro), X (verde) e Y (rojo). Puesto que hay dos señales correspondientes a las sondas de 13, 18 y 21, y una sola señal de cromosomas X e Y, este feto es un varón y es normal con respecto a la prueba de aneuploidía.
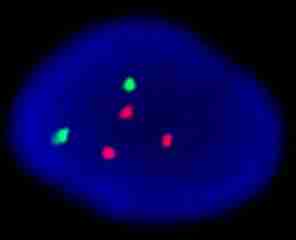
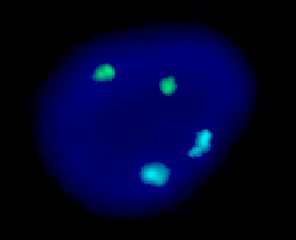
Esta otra prueba de aneuploidía muestra células de un feto femenino con trisomía 21. El núcleo de la izquierda se ha hibridado con sondas diseñadas para los cromosomas 13 (verde) y 21 (rojo), y muestra claramente tres señales rojas. El núcleo de la derecha se ha hibridado con sondas diseñadas para los cromosomas 18 (azul claro), X (verde) e Y (rojo). Puesto que muestra dos señales verdes y ninguna roja, es femenino.