23.1 Mutaciones en la secuencia del DNA inducidas por alteraciones de las bases
Sustitución por desaminación
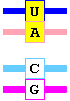
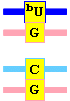
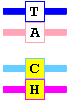
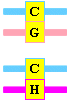
La desaminación oxidativa de la citosina (pág. 302 y 391) la convierte en uracilo, lo que termina introduciendo un cambio de G por A; aunque los sistemas de reparación de errores actúen corrigiendo el U, si lo hacen después de que haya tenido lugar una replicación, la mutación quedará fijada: el par CG pasará a ser un par TA.
![]()
![]()
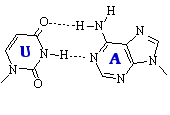
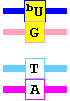
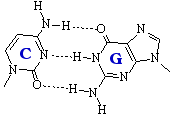
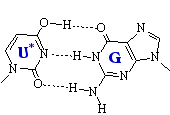
La desaminación oxidativa de la adenina (pág. 302 y 391) la convierte en hipoxantina (esta misma transformación puede ser también el resultado de una hidrólisis ácida). La hipoxantina empareja con citosina (o, más correctamente, se emparejan sus nucleósidos inosina y citidina respectivamente). Como resultado, tras la replicación y aunque se corrija la hipoxantina, aparece un par CG donde había un TA.
![]() desaminación
desaminación
![]() tras replicación
tras replicación
 tras replicación
tras replicación
![]()
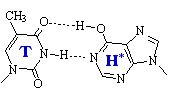
![]()
Sustitución por tautomería
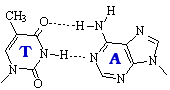
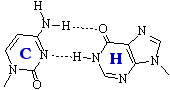
Las formas tautoméricas menos abundantes establecen puentes de hidrógeno diferentes. Por ejemplo, la forma imina de la adenina empareja mejor con citosina. Una vez incorporada ésta, queda fijada la mutación TA→CG aunque más tarde la adenina revierta a la forma amina.
![]() tautomería
tautomería
![]() tras replicación
tras replicación
 tras replicación
tras replicación
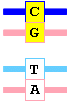
Deleción por despurinación
La rotura del enlace N-glicosídico con una purina deja un sitio abásico (apurínico, pág. 392). De no ser corregido, en la replicación siguiente es probable que se omita esa posición, generando la deleción de un par de bases AT.
![]() despurinación
despurinación
![]() tras replicación
tras replicación
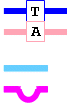
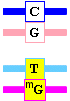
Sustitución por alquilación
La metilación de la guanina por agentes químicos (pág. 393) conduce a O6-metilguanina, que no empareja con citosina, sino con timina. Aunque se elimine más tarde el grupo metilo, la replicación fija la mutación CG→TA.
![]() metilación
metilación
![]() tras replicación
tras replicación
 tras replicación
tras replicación
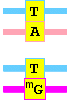
De forma análoga afecta la metilación de la timina a O4-metiltimina, que se empareja con guanina.
![]() metilación
metilación
![]() tras replicación
tras replicación
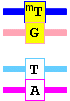 tras replicación
tras replicación
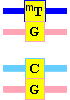
Sustitución por incorporación de bromodesoxiuridina
La adición de bromodesoxiuridina a células en cultivo es una técnica habitual para marcar el DNA y medir la proliferación celular; el fundamento es la analogía estructural de este nucleósido con la timidina. Aparte de esto, el bromouracilo tiene mayor facilidad para adoptar la forma enol que la timidina y la uridina, y dicha forma enol empareja mejor con guanina, introduciendo así mutaciones TA→CG.