Versiones en PDF: ![]() preguntas
preguntas ![]() respuestas
respuestas
Reacciones de la DNA polimerasa I de Escherichia coli.
Si se añade DNA polimerasa I purificada a una mezcla de reacción de pH tamponado que contenga Mg2+, DNA y los cuatro desoxirribonucleósidos-5'-trifosfato marcados con 32P en la posición α (es decir, junto a la desoxirribosa), y se incuba a 37ºC, el 32P pasa a ser insoluble en ácido tricloroacético (TCA) al 10% y puede recogerse en un filtro de membrana –demostrando la formación de DNA, puesto que éste es insoluble en TCA, a diferencia de los nucleótidos. Obsérvese que el DNA de nueva síntesis puede distinguirse del DNA original debido a la radiactividad del primero. Si sólo están presentes tres de los desoxirribonucleósidos-5'-trifosfato, o faltan los iones Mg2+, o el DNA molde, o se añaden ribonucleótidos en lugar de desoxirribonucleótidos, el 32P permanece soluble. Por lo tanto, la polimerización requiere los cuatro desoxirribonucleótidos, iones Mg2+ y DNA. Igualmente, si uno cualquiera de los trifosfatos es reemplazado por el monofosfato o difosfato correspondiente, el 32P permanece soluble (es decir, no hay polimerización). Por consiguiente, el grupo trifosfato es necesario en el sustrato. Si los desoxirribonucleósidos-5'-trifosfato llevan el 32P en posiciones β o γ y 14C en la unidad de desoxirribosa, el 14C queda insoluble y el 32P soluble (puede distinguirse entre 14C y 32P mediante recuento por centelleo líquido). Por lo tanto, los grupos fosfato beta y gamma se eliminan en la reacción y sólo el fosfato α permanece en el polímero. De esta forma, mediante la utilización de radiactividad, pueden distinguirse los diferentes átomos de fosfato.
Masa de una molécula de DNA por marcado del grupo terminal.
La enzima polinucleótido quinasa de Escherichia coli transfiere el grupo fosfato gamma de la adenosina-5'-trifosfato (ATP) al extremo hidroxilo-5' del DNA. Por otra parte, el DNA contiene dos extremos fosforilo-5' (uno en cada cadena polinucleotídica), que pueden convertirse en grupos hidroxilo-5' mediante la acción de la enzima fosfatasa alcalina. En un laboratorio se hicieron reaccionar, mediante la polinucleótido quinasa, 4,7 μg de DNA del fago λ, purificado y homogéneo (o sea, con todas las moléculas del mismo tamaño), que presentaba extremos hidroxilo-5', con [γ-32P]ATP de una actividad específica de 3 mCi/μmol. La muestra se precipitó con ácido y se contó, dando un valor de 1870 cpm en un contador que detectaba el 32P con un 85% de eficiencia. Por consiguiente, se precipitaron 1870 / 0,85 = 2200 dpm de 32P, que equivalen a 2200 / (2,2 x 106) = 0,001 μCi = 1 nCi. Teniendo en cuenta la actividad específica, esto corresponde a 3,3 x 10-7 μmol de ATP, o sea, 1,98 x 1011 moléculas. Como se añadieron 4,7 μg de DNA del fago λ, la masa de una molécula de este DNA será de 4,7 / 9,9 x 1010 = 4,7 x 10-11 μg, o sea, 31,0 x 106 unidades de masa atómica = 31000 kDa. Aquí la radiactividad ha servido para medir la fosforilación de los grupos hidroxilo-5' con una sensibilidad muy elevada. Con este método también pueden distinguirse los grupos fosforilo terminales de nueva formación, con respecto a los antiguos.
Identificación de tirosinas «enmascaradas» en una proteína hipotética.
La tirosina es el único aminoácido que puede ser yodado. En una proteína hipotética se sabe, por el análisis de los aminoácidos, que existen seis residuos de tirosina. Cuando 1 μg de proteína purificada reacciona con 131I, tras precipitación con ácido tricloroacético y recogida en un filtro de fibra de vidrio se detectan 2550 cpm. Si la reacción tiene lugar en presencia de un agente desnaturalizante -de modo que la proteína esté totalmente desplegada- en el precipitado insoluble en ácido se detectan 3820 cpm. Por lo tanto, (2550 / 3820) x 6 =~= 4 tirosinas que pueden yodarse cuando la proteína presenta su configuración nativa. En consecuencia, 6 - 4 = 2 tirosinas que no se yodan y, por tanto, están «enmascaradas» en la estructura tridimensional. En este caso, la radiactividad permite la detección del yodo en la proteína en unas condiciones en que la cantidad de yodo es demasiado pequeña para su detección química.
Permeabilidad de una bacteria al ácido adenílico (AMP).
No todas las sustancias químicas radiactivas que se añaden a un cultivo bacteriano pueden entrar en las bacterias; algunas son excluidas. Si una sustancia radiactiva penetra en la bacteria y se recogen dichas bacterias por filtración, la radiactividad permanecerá en el filtro, Si es excluida, la radiactividad, soluble, pasará a través del filtro y cualquier residuo será eliminado por lavado. Por ejemplo, si se añade [32P]AMP o [3H]AMP (con 3H en el anillo purínico) a un cultivo de Escherichia coli y después de un cierto período de incubación se recogen las bacterias en un filtro de membrana y se lavan con un tampón, no se encuentra en el filtro ni 32P ni 3H. Por lo tanto, las bacterias no incorporan el AMP. En este caso la radiactividad permite distinguir el AMP añadido del AMP presente previamente en la célula. Si se incorporase una cantidad igual al 0,1% de la cantidad ya presente dentro de la bacteria, el aumento sería indetectable por métodos químicos; sin embargo, mediante la utilización de material radiactivo, el 0,1% se puede detectar debido a que el material interno de la célula no está marcado.
Como se están ensayando a la vez [32P]AMP y [3H]AMP, que no se capte ninguno de los dos marcadores indica que la célula no hidroliza el AMP exógeno en adenosina más fosfato, puesto que se sabe que estas dos sustancias pueden entrar en la célula. Ésta es una puntualización de interés debido a que hay muchos ejemplos, como la timidina, en que antes de su entrada en la célula tiene lugar una rotura (dando lugar, en este caso, a timina).
Identificación de una sustancia por precipitación con un anticuerpo.
Si un cultivo de Escherichia coli, multiplicándose en un medio que contiene como única fuente de carbono [14C]glucosa, es infectado con el fago T4 y se permite que continúen los procesos de síntesis en las células infectadas, se sintetizan proteínas codificadas por el genoma del fago. Si se lisan las células infectadas y se añaden anticuerpos específicos contra las proteínas de la cola de T4, se observa 14C en el precipitado. Si se lisan las células en diferentes momentos, a partir de la cantidad de 14C precipitado se puede determinar la velocidad de síntesis de las proteínas víricas de la cola. Puede hacerse un análisis similar con células infectadas por el fago T5 y anticuerpos contra las proteínas de su cola. Sin embargo, si las células son infectadas simultáneamente con T4 y T5 y se lisan, se consigue precipitar el 14C usando el anticuerpo anti-T4, pero no con el anti-T5. Por lo tanto, las proteínas de la cola de T5 no se están sintetizando en la infección mixta. Un experimento como éste es prácticamente imposible sin radiactividad, debido a las pequeñas cantidades que se manejan y a la dificultad en la identificación de las proteínas de la cola en una mezcla tan compleja como es un lisado celular más antisuero.
Purificación de una proteína para la que no existen ensayos biológicos o químicos.
En general, las proteínas se detectan en virtud de una actividad biológica medible (por ej., enzimática, o inhibitoria de alguna enzima) o por una propiedad física distintiva (como la absorción de la luz por la hemoglobina). Sin embargo, no siempre es posible la detección mediante este tipo de procesos y en esos casos se utiliza el método de marcado doble.
Si una bacteria infectada por un fago, capaz de fabricar una proteína determinada, se multiplica en un medio de cultivo que contiene [3H]leucina, todas las proteínas resultarán marcadas con 3H. Si se hace lo mismo con una bacteria infectada con un fago mutante que fabrica sólo un fragmento de la proteína, todas las proteínas resultarán marcadas, pero no habrá proteína intacta que interesa marcada con 14C. Si se mezclan los dos tipos de cultivo marcados, se aíslan sus proteínas y se fraccionan por cromatografía, la mayoría de las fracciones tendrán una misma relación 3H/14C, excepto la proteína de interés, que se encontrará en la fracción que tenga una razón 3H/14C más alta. Por lo tanto, los esquemas de purificación pueden basarse en conseguir un valor máximo de esta razón hasta que se obtenga una fracción que contenga 3H, pero no 14C. Si se habían marcado todas las proteínas, esa fracción contendrá exclusivamente la proteína de interés.
Estudios de reacciones de intercambio.
Como las reacciones de intercambio producen una sustancia químicamente idéntica al material inicial, el análisis químico convencional no puede proporcionar evidencias de la reacción. Sin embargo, el intercambio -o, de modo más general, la participación- se estudia fácilmente con isótopos radiactivos, como puede verse en los dos ejemplos que siguen.
El fosfato de piridoxal es un cofactor en muchas reacciones enzimáticas. Si se utiliza fosfato de [3H]piridoxal con el 3H en determinadas posiciones, se encuentra que el tritio aparece en el agua (3H2O). El fosfato de piridoxal marcado y unido a la enzima puede separarse del agua mediante cromatografía de exclusión en gel y el agua tritiada se puede contar a continuación introduciéndola directamente en una mezcla de centelleo que acepte agua. Si se encuentra 3H en el agua, es una evidencia directa de la participación del fosfato de piridoxal en la reacción mediante la transferencia de un protón.
El DNA es un polinucleótido dicatenario que se mantiene unido por puentes de hidrógeno. Si se añade DNA a agua marcada 3H2O y se extrae después en momentos sucesivos empleando cromatografía de exclusión en gel, aparece 3H en el DNA. A partir de la cinética de intercambio y los efectos de varios agentes desnaturalizantes, puede demostrarse que los puentes de hidrógeno están rompiéndose y volviéndose a formar constantemente. Este fenómeno recibe el nombre de «respiración» de las bases nitrogenadas y un efecto análogo se ha usado también para determinar la velocidad de plegamiento y desplegamiento de. proteínas.
Identificación del lugar activo de las enzimas.
Los isótopos radiactivos pueden jugar un papel de importancia en la identificación de los centros activos de las enzimas, debido a que las reacciones enzimáticas requieren una concentración de enzima tan baja que se necesitarían grandes volúmenes para un análisis químico del tipo tradicional. El siguiente párrafo da un ejemplo sencillo de la determinación del número de sitios de unión.
Puede demostrarse con facilidad que el 1-amino-2-bromoetano se une covalentemente al centro activo de la aminooxidasa del plasma bovino si se marca el segundo átomo de carbono con 14C. La medida de la relación molar entre 14C unido y enzima determina el número de sitios de unión. Hidrolizando la proteína hasta aminoácidos e identificando el tipo de enlace químico puede identificarse el centro de unión (es decir, el -o los- aminoácido que participa en él). Si se conoce la secuencia de aminoácidos de la enzima en cuestión, la hidrólisis parcial en péptidos identificará la localización del aminoácido en la proteína.
Una muestra de ácido [14C]α-cetoglutárico sufre las transformaciones químicas expuestas en el esquema siguiente, en el que se incluyen las actividades especificas en cpm/μmol del CO2 desprendido en cada paso. Determínese el marcaje isotópico del compuesto estudiado. (Para determinar la actividad del CO2 desprendido, éste se precipita como BaCO3.)
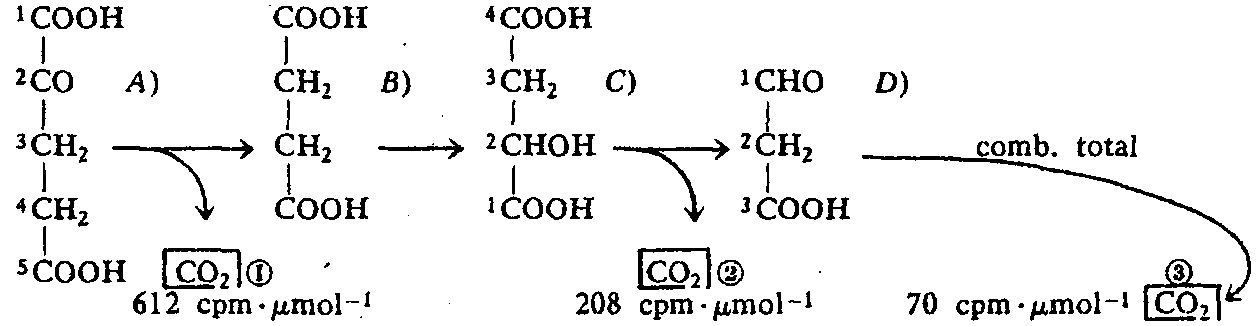
CO2 (1) proviene del carbono 1 de A).
CO2 (2) proviene del carbono 1 de C).
El compuesto C) proviene de B), que presenta simetría molecular, por lo que la actividad de los carbonos simétricos ha de ser idéntica, ya que químicamente son indistinguibles. En consecuencia, el carbono 4 de C) posee la misma actividad que el carbono 1, igual a 208 cpm/μmol.
SD = 3 x 70 cpm/μmol = 210 cpm/μmol
El carbono 3 de D) proviene del carbono 4 de C), que posee. 208 cpm/μmol. Por tanto, todo el marcaje de D) está en el carbono 3. En resumen, el marcaje en el compuesto A) es:
![]()
Merced a la simetría molecular de B), otras posibilidades serían:
a) C-l (612 cpm/μmol), C-2 (418 cpm/μmol).
b) C-l (612 cpm/μmol), C-5 (418 cpm/μmol).
La actividad específica de la timidina marcada con 3H en el radical metilo es de 6 Ci/mmol. ¿Qué fracción de las moléculas de timidina es radiactiva?. Si se marca con esta timidina un DNA de masa molecular 25 x 106 y si el 50% de sus pares de bases son AT, ¿cuántos núcleos de 3H hay por molécula de DNA? Si se dispone de un contador con un 52% de eficacia, ¿qué masa de este DNA daría 1000 cpm?
Si se pudieran utilizar con la misma sencillez 3H, 14C y 32P en un experimento de marcado doble, ¿qué par de isótopos se escogería? ¿Por qué?
En un experimento para medir la síntesis de DNA, ¿qué sería preferible, utilizar [3H]timidina o [32P]timidina? ¿Por qué?
Un contador de centelleo está ajustado de forma que la relación de cuentas entre los canales A y B es de 1000 para el 3H y de 0,2 para el 14C. Se cuenta una mezcla, usando los mismos niveles, rindiendo 1450 cpm en el canal A y 1620 en el canal B. ¿Cuál es la relación de 3H con respecto a 14C en esa muestra?
Haciendo referencia al problema 13, si la muestra contiene [3H]timidina a 6 Ci/mmol y [14C]uracilo a 0,5 Ci/mmol y las eficiencias de recuento son de un 25% para el 3H y de un 82% para el 14C, ¿cuál es la relación molar de timidina y uridina en la muestra? Supóngase que la timidina pertenece a un DNA con un 43% de contenido en GC y la uridina a un RNA con un contenido de un 28% en U; ¿qué masa de RNA y DNA hay en la muestra?
Supóngase que se dispone de un contador Geiger con una eficiencia de un 22% para el 14C y un ruido de fondo de 6 cpm, y de un contador de centelleo con una eficiencia del 72% para el 14C y un ruido de fondo de 38 cpm. En un experimento con 14C se espera que la muestra tenga una actividad muy baja -es decir, de 75 a 100 cpm. ¿Qué contador debería utilizarse? ¿Por qué?
Calcúlese la constante de desintegración del 35S teniendo en cuenta que su periodo de semidesintegración es de 87,1 días.
El 32P tiene un semiperiodo de 14,3 días. Determínese el valor de su constante de desintegración y el porcentaje inicial que queda al cabo de 15, 25 y 50 días en una muestra de este radioisótopo.
En un contador Geiger se ha determinado la actividad de una muestra de 35S en diferentes días. A partir de los valores obtenidos, determínense el periodo de semidesintegración y la constante de desintegración de este radioisótopo:
| Tiempo (días) | 0 | 1 | 2 | 3 | 4 | 5 | 10 | 15 |
| cpm | 4290 | 4247 | 4210 | 4180 | 4136 | 4110 | 3952 | 3796 |
Calcúlese el número de átomos radiactivos y la masa en gramos de 1 Ci de 32P.
¿Cuál será la actividad específica máxima de la [14C]glucosa? (Masa molecular: 192; semiperiodo del 14C: 5700 años)
Calcúlese la actividad específica máxima de la [14C]valina en Ci/g
¿Cuál será la máxima actividad específica teórica, en Ci/mmol, con la que se podría preparar uniformemente marcada la [14C]tirosina? ¿Qué proporción de moléculas están realmente marcadas en una preparación de [14C]tirosina que posee una actividad específica de 250 mCi/mmol?
Con objeto de determinar el volumen de sangre de un ave se le inyectaron 2 ml de una suspensión de eritrocitos marcados radiactivamente con una actividad de 1,77 x 107 cpm/ml. Tras cinco minutos se aisló una pequeña muestra de sangre del animal, obteniéndose en ella una actividad de 1,5 x 105 cpm/ml. Determínese el volumen sanguíneo del ave estudiada.
Un cultivo de un microorganismo se incubó en presencia de [14C]dATP (1 nCi/μmol), aislándose a continuación el DNA de dicho microorganismo. Por tratamiento de este ácido nucleico con endonucleasas se obtuvo un polinucleótido cuya composición cuantitativa de bases es A3C4G3T4. ¿Cuál será la actividad específica de dicho polinucleótido? (El tiempo de incubación fue suficiente para obtener todo el DNA uniformemente marcado.)
1 ml de una disolución acuosa de [14C]uridina tiene una actividad especifica de 200 Ci/mol y una actividad total de 50 μCi. ¿Cuántos mililitros de una disolución de uridina fría 5 mM y cuántos de agua es necesario añadir a la de uridina marcada para obtener una disolución de concentración 2 mM y actividad especifica 5 Ci/mol? Descríbase la preparación de 1 ml de disolución de uridina 1 mM y actividad especifica 2 Ci/mol.
Para valorar la cantidad de glicina presente en un hidrolizado de proteína se tomaron 152,6 mg de éste y se mezclaron con 6,0 mg de [14C]glicina con una actividad específica de 120 cpm/mg. Después de aislar parte de la glicina y purificarla totalmente, se encontró que su actividad específica era de 59 cpm/mg. Determínese la proporción de glicina que había en el hidrolizado inicial.
Una única cadena de DNA (+) de composición de bases 19% A, 31% G, 31% C y 19% T se incuba en presencia de DNA polimerasa y [14C]dATP (1 mCi/mol), obteniéndose la cadena complementaria (-). El DNA de doble hélice obtenido es utilizado como molde por la RNA polimerasa, en presencia de [14C]CTP (2 x 106 cpm/g), la cual transcribe la cadena (-). ¿Cuál será la actividad específica del DNA dicatenario y del RNA obtenido en la transcripción? La eficacia en todos los recuentos es del 50%.
En la determinación del aminoácido N-terminal de una proteína que consta de dos cadenas polipeptídicas idénticas se utilizó cloruro de [14C]dansilo (0,1 Ci/mol). La reacción se llevó a cabo con 100 nmol de proteína. Mediante cromatografía se aisló el dansilaminoácido N-terminal en un volumen de 2 ml. Una alícuota de 0,1 ml de éste proporcionó una actividad de 1385 cpm en un contador de centelleo líquido. Calcúlese el rendimiento de la reacción, sabiendo que la eficacia del recuento es del 80%.
Por el método de síntesis en fase sólida de Merrifield se ha tratado de sintetizar el péptido Gly-Val-Pro-Ala a base de [14C]glicina. Debido a un mal acoplamiento con la resina se han originado en el proceso dos péptidos cuya composición de aminoácidos es: 1) Gly, Val, Ala, Pro, y 2) Gly2, Pro, Ala. El péptido que se trataba de sintetizar se ha obtenido con una pureza del 90%. ¿Cuál será la actividad específica de la mezcla de péptidos obtenidos, en Ci/g?. La actividad específica de la [14C]glicina utilizada es 104 cpm/mmol. La eficacia del recuento es del 80%. La masa molecular media para todos los aminoácidos considérese 110.
Determínese el número de átomos de 131I y 32P que dan lugar a 1 dpm, sabiendo que los periodos de semidesintegración son, respectivamente, de 8,1 y 14,3 días.
Para la carboximetilación de una proteína se ha utilizado yodo[14C]acetato (1 mCi/mmol). Teniendo en cuenta que la proteína contiene cinco residuos de cisteína y que la reacción precisa un exceso dos veces molar de yodoacetato, ¿cuántos mCi de yodo[14C]acetato se precisarán para carboximetilar 1 nmol de proteína? ¿Cuál será la actividad específica de la proteína carboximetilada? Considérese que en la reacción sólo se modifican las cisteínas.