Respuestas:
Asequibles, debe saberse resolverlos:
Algo más difíciles:
| 10 |
11 |
12 |
13 |
14 |
| 15 |
16 |
17 |
18 |
19 |
| 20 |
21 |
22 |
23 |
24 |
| 25 |
26 |
27 |
28 |
29 |
| 30 |
31 |
A continuación se pone ayuda para resolver algunos de ellos, y luego las respuestas.
Respuestas: Asequibles, debe saberse resolverlos:
|
|
Para el cálculo de la segunda pregunta se necesita el valor de masa molecular de un nucleótido:
| adenina: 135 | desoxiadenosina: 251 | desoxiadenílico: 331 |
| guanina: 151 | desoxiguanosina: 267 | desoxiguanílico: 347 |
| timina: 126 | desoxitimidina: 242 | desoxitimidílico: 322 |
| citosina: 111 | desoxicitidina: 227 | desoxicitidílico: 307 |
| masa media de un desoxinucleótido: 327 | ||
| masa media de un nucleótido formando parte de un DNA: 327-18=309 | ||
| masa media de un par de bases: 309 x 2 = 618 |
Por lo tanto, una molécula de DNA de 25 Mpb tiene:
25 x 106 / 618 = 40450 pb
A partir de ahí se calcula el número de timinas.
Respuesta completa![]()
Piensa en qué otras moléculas pueden resultar marcadas. Respuesta
completa![]()
Un proceso de desintegración radiactiva debe seguir la ecuación N = N0 exp(-λt), o dN/dt = -λN. Se ha medido la radiactividad (cpm), que corresponde a -dN/dt, es decir, debe seguir la ecuación A = λN0 exp(-λt).
Para obtener el valor de la constante de desintegración λ debemos ajustar los datos a esta ecuación: lo mejor es por regresión no lineal; en su defecto, tomando logaritmos: ln(A) = ln(λN0) - λt obtenemos una recta, que se puede ajustar por mínimos cuadrados, cuya pendiente es -λ.
El tiempo de semidesintegración se relaciona con la constante de desintegración por: t1/2 = ln2 / λ
La "actividad específica máxima" es la actividad específica (es decir, actividad o velocidad de desintegración, por unidad de masa o por mol) que hay cuando todos los átomos aludidos están como su isótopo radiactivo.
Por ejemplo:
Respuestas completas: 20![]() ,
21
,
21![]() ,
22
,
22![]()
El volumen de sangre del organismo se determina a través del cálculo de la dilución.
Si el DNA se ha marcado uniformemente, quiere decir que cualquier región de la molécula tendrá sus residuos de adenosina marcados en igual medida. Las endonucleasas producen un fragmento de la molécula, cuya composición se indica. Ésta nos indica el número de adenosinas y, por tanto, la radiactividad.
El adjetivo "frío" se usa, en el contexto del marcaje radiactivo, para indicar el compuesto que no está marcado.
Aplicar al proceso de mezcla los balances de masa y de radiactividad.
a) Los signos (+) y (-) se utilizan para designar las hebras complementarias (respectivamente, hebra con sentido y hebra antisentido). En nuestro caso, basta saber que la hebra que sintetiza la DNApol es complementaria a la de partida, y por tanto tendrá A donde hubiera T, T donde hubiera A, C donde hubiera G y G donde hubiera C. La RNApol sintetizará una hebra de RNA con A donde hubiera T, U donde hubiera A, C donde hubiera G y G donde hubiera C:
| DNA (+) | A | G | C | T |
| DNA(-) | T | C | G | A |
| RNA(+) | A | G | C | U |
b) Se necesita la masa molecular del CTP. La citidina tiene 227, el
ácido citidílico o CMP tiene 307, cada fosfato añadido
supone 80 (véase la ayuda de la pregunta 10![]() );
por lo tanto, el CTP tiene 307 +80 +80 = 467.
);
por lo tanto, el CTP tiene 307 +80 +80 = 467.
1 mmol equivale a 6.023·1020 moléculas = 6.023·1020
grupos metilo
6 Ci = 6 x 2.22·1012 dpm
t1/2(3H) = 12.3 años, λ
= ln2 / 12.3 años
Radiactividad = dN / dt = 6 x 2.22·1012 dpm = ln2
/ 12.3 años x 6.023·1020 x f
|
|
|
| f = | |
|
|
f = 0.206 es decir, el 20.6% de las moléculas son radiactivas
(3H).
(léase la ayuda de arriba![]() )
)
Por lo tanto, una molécula de DNA de 25 Mb tiene: 25 x 106
/ 618 = 40450 pb
A partir de ahí se calcula el número de timinas: 40450
/ 2 = 20224
(pues la mitad de los pares de bases son AT y el
número de T es igual al número de pares AT).
De las cuales el 20.6% son radiactivas: 20224 x 0.206 = 4166 átomos
de 3H por molécula de DNA
|
|
|
||
| Actividad = λ N = | | x 4166 x | |
|
|
|
luego mDNA = 2.58·10-13 g/min x A
|
|
||
| A = | | = 1923 dpm, por tanto mDNA = 4.96·10-10 g = 0.496 ng = 496 pg |
|
|
Aquéllos cuyos espectros de emisión se solapan menos: 3H y 32P.
La timidina sólo se incorpora al DNA, mientras que el fosfato se incorporará también a RNA, fosfoproteínas y fosfolípidos. Por ello, es preferible la timidina, por ser más específica, independientemente del isótopo.
| cpm 3H }A | cpm 14C }A | |||
| | = 1000 | | = 0.2 | |
| cpm 3H }B | cpm 14C }B |
cpm}A = 1450
cpm}B = 1620 suman 3070 cpm
3H en B es 0.001 = 0.1% del 3H total: despreciable
(el máximo sería de 0.001 x 1450 = 1.4 cpm)
Por tanto, cpm 3H }B »
0 , y cpm 14C }B = 1620
cpm 14C }A = 0.2 x 1620 = 324 y la suma cpm 14C
}A+B = 1944
cpm 3H }A = 1450 324 = 1126
cpm 3H }B = 676 / 1000 »
0 en efecto y la suma cpm 3H }A+B = 1126
Verificamos la suma 3H + cpm 14C = 1944+1126
= 3070 correcto
Respuesta: la relación tritio/carbono-14 es de 1126/1944 = 0.58
a) Para una relación tritio/carbono-14 de 0.58:
| 0.58 cpm 3H | 100 dpm 3H | 82 cpm 14C | dpm 3H | Ci 3H | ||||
| | x | | x | | = 1.90 | | = 1.90 | |
| 1 cpm 14C | 25 cpm 3H | 100 dpm 14C | dpm 14C | Ci 14C |
| Ci 3H | mmol T | 0.5 Ci 14C | mol T | ||||
| 1.90 | | x | | x | | = 0.158 | |
| Ci 14C | 6 Ci 3H | mmol U | mol U |
b)
Un DNA con un 43% de G+C tiene un (100-43)/2 = 28.5% de T. Es decir,
hay 28.5 moles de T por cada 100 moles de nucleótidos.
La actividad de 3H era 1126 cpm, con lo cual:
| 100 dpm | 1 Ci | mmol T | 100 mmol (nt DNA) | ||||||
| 1126 cpm | x | | x | | x | | x | | = |
| 25 cpm | 2.22·1012 dpm | 6 Ci | 28.5 mmol T |
= 1.19·10-9 mmol = 1.19 pmol de desoxirribonucleótidos
Puesto que nos piden la masa de DNA, necesitamos la masa molecular
media de un desoxirribonucleótido (véase la ayuda del problema
10![]() ),
que es 309 y así calculamos:
),
que es 309 y así calculamos:
1.19 pmol x 309 g/mol = 368 pg de nucleótidos = 368 pg de DNA = 0.368 ng.
En el RNA hay 28 moles de U por cada 100 moles de nucleótidos.
Similarmente, con las 1944 cpm de 14C :
| 100 dpm | 1 Ci | mmol U | 100 mmol (nt DNA) | ||||||
| 1944 cpm | x | | x | | x | | x | | = |
| 82 cpm | 2.22·1012 dpm | 0.5 Ci | 28 mmol T |
= 7.63·10-9 mmol = 7.63 pmol de ribonucleótidos
La masa molecular media de un ribonucleótido es de 309+16=325.
7.63 pmol x 325 g/mol = 2480 pg de nucleótidos = 2480 pg de RNA = 2.48 ng
| Geiger | Centelleo | |||
| muestra | señal | señal/ruido | señal | señal/ruido |
| 75 dpm | 75 x 0.22 = 16.5 cpm | 16.5/6 = 2.75 | 75 x 0.72 = 54 cpm | 54/38 = 1.42 |
| 100 dpm | 100 x 0.22 = 22 cpm | 22/6 = 3.67 | 100 x 0.72 = 72 cpm | 72/38 = 1.89 |
A pesar de que el contador Geiger es menos eficaz (detecta menos cuentas), su relación señal/ruido es mejor, por lo que debe usarse éste.
| ln2 | 1 d | 1 h | ||||
| λ = | | = 0.0080 d-1 x | | x | | = 5.5·10-6 min-1 |
| t1/2 | 24 h | 60 min |
| ln2 | 1 d | 1 h | ||||
| λ = | | = 0.048 d-1 x | | x | | = 3.3·10-5 min-1 |
| t1/2 | 24 h | 60 min |
|
|
||
| "% inicial que queda" = | | x 100 = 100 x e-λ t |
|
|
|
|
|
|
|
|
|
|
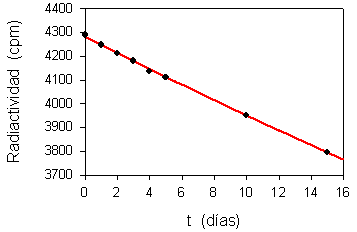
a) Representación directa de los datos y regresión no lineal: Ecuación: Y = a exp(-bX)
|
 |
b) Representación de los datos transformados matemáticamente y regresión lineal: Ecuación: Y = a + b X
|
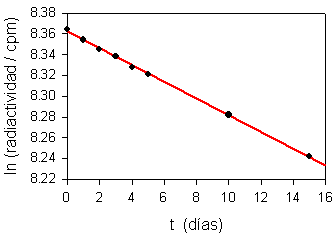 |
(nota: aunque las gráficas parecen iguales, esto sólo es así porque los datos corresponden al inicio de la curva de desintegración; si hubiese datos a tiempos mayores se apreciaría que la de arriba es una curva exponencial y la de abajo es una recta)
1 Ci = 2.22·1012 dpm; t1/2 = 14.2 d
| ln2 | ln2 | ||
| λ = | | = | |
| t1/2 | 14.2 x 24 x 60 min |
| dN | ln2 | |||||
| | = | λ N | 2.22·1012 min-1 = | | x N | |
| dt | 14.2 x 24 x 60 min |
N = 6.55·1016 átomos radiactivos
que pesan 32 u.m.a. cada uno; en total:
|
|
|
| | = 3.48·10-6 g = 3.48 μg de átomos radiactivos |
|
|
(de los átomos de P no radiactivos no podemos afirmar nada, pues el dato de radiactividad no depende de ellos)
El periodo de semidesintegración del 14C es de 5700 años =
5700 a x 365.25 d/a x 24 h/d x
60 min/h = 3.00·109 min
| ln2 | ln2 | |||
| λ = | | = | | = 2.31·10-10 min-1 |
| t1/2 | 3.00·109 min |
La actividad del 14C es:
| dN | ||
| A = | | = λ N = 2.31·10-10 min-1 x N = N x 2.31·10-10 dpm |
| dt |
| 1 Ci | |||
| A = | N x 2.31·10-10 dpm x | | = N x 1.04·10-22 Ci |
| 2.22·1012 dpm |
La glucosa tiene 6 átomos de carbono. Si todos ellos fueran 14C,
el número de átomos radiactivos en un mol de glucosa sería
N = 6 x 6.023·1023, con lo que la actividad
específica máxima es
AE = 6 x 6.023·1023 mol-1
x 1.04·10-22 Ci = 376 Ci/mol
Si la queremos por unidad de masa, usamos la masa molecular de la glucosa:
AE = (376 Ci/mol) / (192 g/mol) = 1.96
Ci/g
La actividad del 14C es: A = N x 1.04·10-22 Ci (véase la respuesta al anterior).
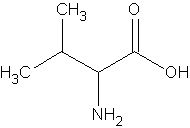 La valina tiene
5 átomos de carbono y una masa molecular de 117. Si todos ellos
fueran 14C, el número de átomos radiactivos en
un mol de valina sería N = 5 x 6.023·1023,
con lo que la actividad específica máxima es
La valina tiene
5 átomos de carbono y una masa molecular de 117. Si todos ellos
fueran 14C, el número de átomos radiactivos en
un mol de valina sería N = 5 x 6.023·1023,
con lo que la actividad específica máxima es
AE = 5 x 6.023·1023 mol-1
x 1.04·10-22 Ci = 313 Ci/mol
Y por unidad de masa es:
AE = (313 Ci/mol) / (117 g/mol) = 2.67
Ci/g
(Respuesta simplificada; véanse los detalles en las respuestas de 20 y 21).
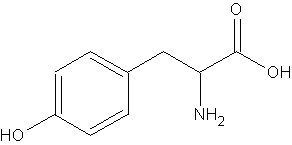 a) La actividad
del 14C es: A = N x 1.04·10-22 Ci
a) La actividad
del 14C es: A = N x 1.04·10-22 Ci
La tirosina tiene 9 átomos de carbono y una masa molecular de
181. La actividad específica máxima será de
AE = 9 x 6.023·1023 mol-1
x 1.04·10-22 Ci = 564 Ci/mol
= 0.564 Ci/mmol
b) 250 mCi/mmol / 564 mCi/mmol = 0.443 = 44.3% de los átomos de C serán 14C.
La actividad (concentración) se diluye en el volumen total de sangre:
Ai x Vi = Af
x Vf
1.77·107 cpm/mL x 2 mL =
1.5·105 cpm/mL x (2mL+V)
V = 234 mL
La actividad del fragmento de DNA será de 1 nCi/mmol por cada residuo de A que contenga, es decir:
AE = 3 nCi/μmol = 3 mCi/mol
| disolución de partida marcada | disolución de partida fría | agua | disolución final |
|
|
|
|
|
| V1 = 1mL
AE1 = 200 Ci/mol A1 = 50 μCi |
V2 mL
C2 = 5 mM |
V3 mL | V4 = V1 + V2
+ V3
C4 = 2 mM AE4 = 5 Ci/mmol |
Balance de materia:
C1 V1 + C2 V2 = C4
V4 ; 0.25 mM x 1 mL + 5 mM x V2 mL = 2 mM x (1 +
V2 + V3 ) mL
Y balance de actividad:
A1 + 0 + 0 = A4 ; 50 μCi
= AE4 x (0.25 μmol
+ 5 μmol/mL
x V2 mL + 0) ; V2 = 1.95 mL
Entre ambas: V3 = 2.05 mL
Respuesta: se debe mezclar la uridina marcada (1 mL) con 1.95 mL de uridina fría y 2.05 mL de agua.
Balance de masa (de Gly):
f x 152.6 mg +
6 mg = m
Balance de radiactividad (de [14C]Gly):
0 + 120 cpm/mg x 6 mg = 59 cpm/mg x m
| ( f es la fracción en masa de Gly,
o riqueza, con respecto al hidrolizado)
( m es la masa de Gly en la mezcla final) |
De la segunda ecuación: m
= 720/59 = 12.20 mg
Que, llevado a la primera ecuación, da: f
= 6.20/152.6 = 0.0406 = 4.06%
Los contenidos (% mol de base/mol de ácido nucleico) de cada hebra serán:
| A | G | C | T | |
| DNA(+) | 19% | 31% | 31% | 19% |
| DNA(-) | 19%(*) | 31% | 31% | 19% |
| U | ||||
| RNA(+) | 19% | 31% | 31% | 19%(*) |
Como sólo una de las dos hebras está marcada, el DNA de doble hebra tiene un 9.5% de adenosina radiactiva, mientras que el RNA tiene un 19% de citidina radiactiva.
a) Actividad del DNA:
1 mol de dsDNA (double strand DNA, DNA bicatenario) tiene
una actividad:
0.095 mol A* x 1 mCi/mol = 0.095 mCi
Luego la actividad específica del dsDNA es de 0.095 mCi/mol
b) Actividad del RNA:
1 mol de RNA tiene una actividad de:
0.31 mol C* x 2·106 cpm/(g CTP)
x (100 dpm/50 cpm) x (1 Ci/2.22·1012
dpm) = 0.559 μCi
x (mol C) / (g CTP)
Como la masa molecular del CTP es de 467 (véase la ayuda de
arribaμ),
su masa molar es 467 g/mol y entonces la actividad es: 0.559 x 467 μCi
= 261 μCi
Y la actividad específica del RNA es de 261 μCi/mol
= 0.261 mCi/mol
Si el rendimiento fuera del 100% se incorporaría 1 mol de dansilo por mol de polipéptido (o de aminoácido N-terminal); a 100 nmol de proteína, con 2 aminoácidos N-terminales, se incorporarían 200 nmol de dansilo.
Veamos los que se han incorporado realmente:
| 1385 cpm | 100 dpm | ||||
| A = | | x 2 mL = 27700 cpm | x | | = 34625 dpm |
| 0.1 mL | 80 cpm |
| 34625 dpm | 1 Ci | ||
| | x | | = 1.56·10-7 mol de dansilo incorporado |
| 0.1 Ci/mol | 2.22·1012 dpm |
Por tanto, el rendimiento de la dansilación es de
| 1.56·10-7 mol | |
| | = 0.78 = 78% |
| 200·10-9 mol |
El péptido 1, que es el deseado (Gly-Val-Pro-Ala), tendrá una actividad de 104 cpm/mmol
El péptido 2, al tener dos residuos de Gly, tendrá una actividad de 2·104 cpm/mmol
La actividad específica de la mezcla será:
AE = 0.90 x 104 cpm/mmol + 0.10 x 2·104 cpm/mmol = 1.1·104 cpm/mmol
Para pasar a μCi/g, teniendo en cuenta que 1 mol de cualquiera de los péptidos tiene 4 moles de aminoácidos, pesará 4 x 110 gramos y:
| 100 dpm | 1 Ci | 103 mmol | 1 mol | ||||
| AE = 1.1·104 cpm/mmol x | | x | | x | | x | |
| 80 cpm | 2.22·1012 dpm | mol | 440 g |
a) Para 131I:
| ln2 | ln2 | |||
| λ = | | = | | = 0.0856 d-1 |
| t1/2 | 8.1 d |
| dN | ||
| 1 dpm = 1 min-1 = A = | | = λ N = 0.0856 d-1 x N |
| dt |
| 1 min-1 | 24 h | 60 min | ||||
| N = | | x | | x | | = 16800 (átomos) |
| 0.0856 d-1 | 1 d | 1 h |
b) Similarmente para 32P: N = 29700 átomos
(una vida media más larga significa una menor radiactividad
para el mismo nº de átomos, por eso para conseguir la misma
actividad hace falta mayor cantidad de P).
"yodoacetato-14C" debe escribirse correctamente como yodo[14C]acetato
La reacción es Cys + 2 IAcO-
| 1 mCi | 5 mol Cys | |||
| AEprot. = | | x | | = 5 mCi/mmol prot. = 5 Ci/mol |
| mmol Cys | mol prot. |