a
c (×12)
δ,ε
γ
α (×3)
β (×3)
b (×2)

ATP sintasa; ATPasa transportadora de protones; EC 7.1.2.2
 (translocasa)
antes EC 3.6.3.14 (hidrolasa)
(translocasa)
antes EC 3.6.3.14 (hidrolasa)
Cataliza la reacción ADP + Pi ![]() ATP + H2O
( → ATP sintasa ; ← ATPasa )
ATP + H2O
( → ATP sintasa ; ← ATPasa )
La acción catalítica de esta sintasa se activa con los cambios conformacionales que se provocan gracias al paso de protones a través de la proteína, impulsados por el gradiente de protones generado durante el transporte electrónico:
| Balance global en la ATP sintasa: | 12 H+intermem./intratilacoidal | 12 H+matriz/estroma | |
| (3 ADP + 3 Pi)matriz/estroma | 3 ATPmatriz/estroma |
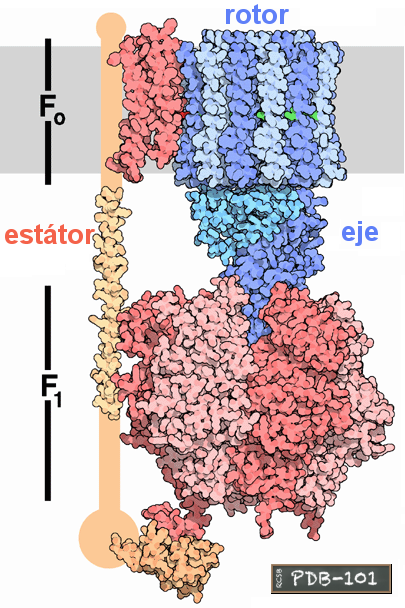
Pasa el puntero sobre la imagen para mostrar la identificación de las subunidades.
La sintasa se llama FoF1 (la del cloroplasto, CFoCF1), al considerarse formada por dos partes o dominios:
La descripción como una combinación de Fo y F1 es demasiado simple; hoy en día se conoce la estructura con más detalle y no es sencillo definir si algunas subunidades pertenecen a Fo o a F1.
![]() Estructura molecular 3D (ampliación)
Estructura molecular 3D (ampliación)
| Las subunidades alfa y beta (3 de cada) alternan entre 3 conformaciones: |
|
Nomenclatura alternativa, en algunos textos: TP = T (tense, tensa) DP = L (loose, laxa, relajada) E = O (open, abierta) |
Además, cada cambio conformacional en un dímero se transmite a los dímeros vecinos. En cada momento, cada uno de los 3 dímeros α β se encuentra en una de las conformaciones:

El cambio conformacional de las subunidades alfa y beta se inicia por un "empujón" que ejerce sobre ellas la subunidad gamma al ir girando. (Gamma, delta y épsilon constituyen el eje del rotor)
El paso de protones, a favor de gradiente, desde el espacio intermembranario mitocondrial hacia la matriz o estroma mitocondrial —o bien desde la luz tilacoidal hacia el estroma cloroplástico— sólo puede hacerse a través de la proteína ATP sintasa. Los protones pasan a través de las subunidades a y c, lo que hace girar el conjunto de subunidades c ("carrusel"), que a su vez hace girar también a delta, épsilon y gamma (el "eje").
|
Rotor: c12 + γ + δ + ε Estátor: a + b2 + (α β)3 |
|

rotor
estátor
|
Para comprender mejor todo esto conviene observar las excelentes imágenes animadas creadas en MRC Mitochondrial Biology Unit, de Cambridge; pulsa sobre cada icono de película
Referencia de la imagen PDB-101: PDB Molecule of the Month - ATP Synthase. David Goodsell, 2005. doi:10.2210/rcsb_pdb/mom_2005_12
Licencia CC-by, 2015 David Goodsell & RCSB Protein Data Bank.
Referencia de la imagen del motor: Commons; autor: Zureks; licencia CC-by-sa.
Referencia de los vídeos: Molecular animations of ATP synthase. John Walker et al., MRC Mitochondrial Biology Unit, Cambridge, UK. https://www.mrc-mbu.cam.ac.uk/research-groups/walker-group/molecular-animations-atp-synthase [consultado el 20 abril 2023] Los vídeos residen en el servidor de YouTube.
Copyright © MRC MBU 2015.