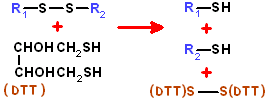Presentación e instrucciones breves
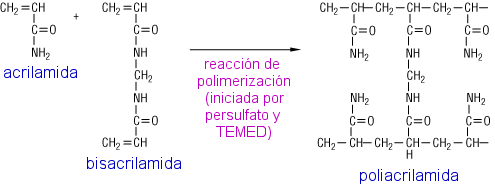
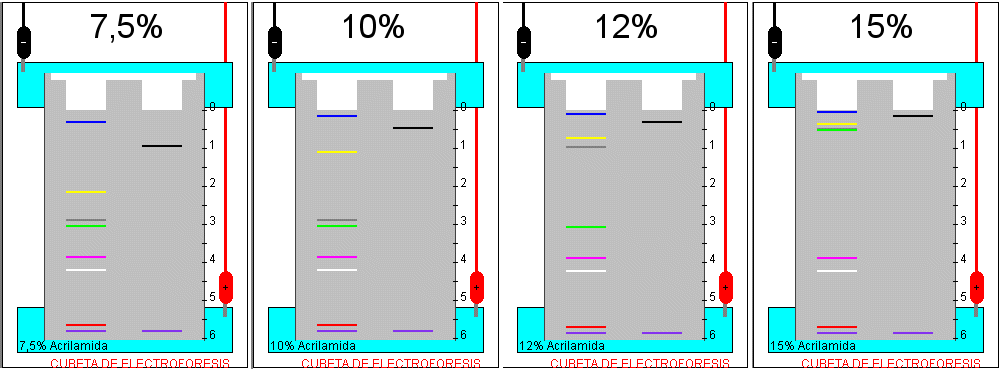
Esta aplicación simula el proceso de electroforesis de las proteínas en un gel de poliacrilamida con SDS. Tras la separación, se puede obtener un gráfica con los resultados. En ésta, se determina la masa molecular experimental de las muestras a partir de su movilidad comparada con la de los patrones.
Puedes aprender más sobre el fundamento de este método de separación en la sección de técnicas.
Brevemente:
- Elige una velocidad para la animación (depende de
tu ordenador)
-
Elige una muestra (Problemas nº 1 a 10).
- Elige la concentración de acrilamida en el gel (entre
7,5% y 15%).
- Elige 2 o más patrones, marcando las casillas que
hay junto a sus nombres. Puedes consultar su identidad y propiedades pulsando
el botón Info. proteínas.
- Elige un voltaje.
- Pulsa el botón Añadir patrón para cargar los patrones
en el primer pocillo.
- Pulsa el botón Añadir muestra para cargar la muestra
en el segundo pocillo.
- Pulsa el botón Comenzar para conectar la corriente
y comenzar la electroforesis. Antes de que las bandas se salgan por abajo,
pulsa el botón Detener para pararla.
Se buscan colaboradores:
para sacar partido a este simulador, sería interesante disponer de ejercicios o guiones que lo utilicen; también serán bienvenidos datos contrastados de otras proteínas.
Ejercicios
A) Influencia de la diferencia de potencial aplicada
Carga en el gel todos los patrones y una cualquiera de las muestras problema de proteínas.
Elige por turno cada una de las diferencias de potencial (voltajes) disponibles en el simulador y desarrolla la electroforesis hasta que la banda del colorante (color morado) se acerque al extermo inferior del gel. Anota en cada caso la movilidad de las bandas de las proteínas patrón.
1. ¿Cuál es el efecto de aumentar la diferencia de potencial eléctrico entre ambos electrodos?
B) Influencia de la concentración de acrilamida
Carga en el gel todos los patrones y una cualquiera de las muestras problema de proteínas.
Elige por turno cada una de las concentraciones de acrilamida disponibles en el simulador y desarrolla la electroforesis hasta que la banda del colorante (color morado) se acerque al extermo inferior del gel. Anota en cada caso la movilidad de las bandas de las proteínas patrón.
1. ¿Cuál es el efecto de aumentar la concentración de acrilamida que forma el gel?
2. ¿Cómo lo interpretas, en términos del mecanismo de la separación?
C) Autoevaluación de los resultados de masa molecular
Elige una de las muestras problema de proteínas, incluidas en el simulador.
Carga esa muestra en el gel, así como todos los patrones.
Desarrolla la electroforesis hasta que la banda del colorante (color morado) se acerque al extermo inferior del gel.
Tras acabar, mide la movilidad de la proteína problema e interpola ese valor en la gráfica para obtener su masa molecular relativa.
Para confirmar que tus resultados son correctos:
| |
escribe aquí tu resultado de masa molecular relativa |
| Muestra problema nº 1 |
|
| Muestra problema nº 2 |
|
| Muestra problema nº 3 |
|
| Muestra problema nº 4 |
|
| Muestra problema nº 5 |
|
| Muestra problema nº 6 |
|
| Muestra problema nº 7 |
|
| Muestra problema nº 8 |
|
| Muestra problema nº 9 |
|
| Muestra problema nº 10 |
|
D) Determinación de estructura cuaternaria
Carga en el gel del simulador todos los patrones y una cualquiera de las muestras problema de proteínas (aunque no la usarás).
Desarrolla la electroforesis hasta que la banda del colorante (color morado) se acerque al extermo inferior del gel.
Tras acabar, accede a la gráfica de masa molecular relativa frente a movilidad y resuelve los siguientes problemas:
1. Una enzima tratada con SDS y beta-mercaptoetanol se analiza en un gel de poliacrilamida con SDS, observándose 2 bandas con una movilidad relativa de 0,47 y 0,59.
Gracias a experimentos independientes, usando cromatografía de exclusión y ultracentrifugación, se ha determinado que la masa molecular de la enzima nativa es de 128 000.
Empleando la recta de calibrado obtenida en el simulador, estima la masa molecular de ambas subunidades de la enzima y deduce cuál es su estructura cuaternaria.
2. Una proteína tratada con SDS pero sin beta-mercaptoetanol se analiza en un gel de poliacrilamida con SDS, observándose una sola banda con una movilidad relativa de 0,21.
Cuando la proteína se trata previamente con SDS y beta-mercaptoetanol, la electroforesis muestra dos bandas, de movilidades 0,48 y 0,61.
Empleando la recta de calibrado obtenida en el simulador, interpreta los resultados.
3. (Avanzado) Si la proteína se trata previamente con tripsina, se obtiene la misma banda en ausencia de beta-mercaptoetanol, pero cuatro bandas en su presencia. Explica estos resultados.
Angel Herráez. Parte de la sede web Biomodel.uah.es
❖
This page is also available in English.
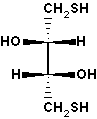 treo-2,3-dihidroxi-1,4-butanoditiol. También llamado reactivo de Cleland.
treo-2,3-dihidroxi-1,4-butanoditiol. También llamado reactivo de Cleland.