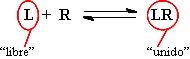
Típicamente se llama ligando a la molécula pequeña y receptor a la proteína, pero su papel es intercambiable. En general, se trabaja con el ligando purificado, mientras que el receptor forma parte de una muestra compleja, y se varía el ligando para determinar la presencia, cantidad y características del receptor.
Se trata como un equilibrio químico:

R = receptor (estrictamente, sitio de unión en un receptor)
L = ligando
[x] = concentración molar de x
B = concentración de ligando unido (bound)
F = concentración de ligando libre, es decir, no unido (free)
T = concentración total de ligando = B + F
Bmáx = unión máxima = concentración total de receptor (estrictamente, concentración de sitios de unión; esta diferencia es relevante en el caso de que una molécula de receptor tenga varios sitios o centros de unión para el mismo ligando).
L* = ligando marcado, con una actividad específica conocida. Generalmente con un isótopo radiactivo (radioligando), pero es aplicable a cualquier otro tipo de marcaje siempre que no afecte a la unión.
L° = ligando no marcado (“frío”).
Un ensayo de unión puede ser:
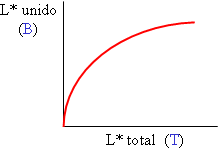 Un ensayo de saturación (o curva de unión): se mide la cantidad de ligando unida al variar la cantidad total añadida, para una cantidad fija de receptor. El ligando está marcado (L*) y se mide el resultado tras alcanzar el equilibrio.
Un ensayo de saturación (o curva de unión): se mide la cantidad de ligando unida al variar la cantidad total añadida, para una cantidad fija de receptor. El ligando está marcado (L*) y se mide el resultado tras alcanzar el equilibrio.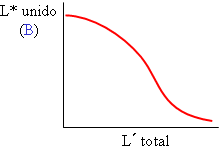 Un ensayo de desplazamiento (o de competencia): se mide la cantidad de ligando marcado unida al variar la cantidad de un ligando competidor, que puede ser el mismo ligando sin marcar (L°) o bien otra molécula diferente (L´) que también se una al receptor en el mismo sitio. La cantidad de receptor se mantiene constante. También se mide a equilibrio.
Un ensayo de desplazamiento (o de competencia): se mide la cantidad de ligando marcado unida al variar la cantidad de un ligando competidor, que puede ser el mismo ligando sin marcar (L°) o bien otra molécula diferente (L´) que también se una al receptor en el mismo sitio. La cantidad de receptor se mantiene constante. También se mide a equilibrio.En cualquiera de los casos, se analiza exclusivamente el ligando detectable, es decir, el que está marcado, pero el equilibrio o cinética se rige por la suma de ligando marcado L* y no marcado L°, “no distingue” entre ambos, la identidad química de ambos es la misma.
A partir de la anterior ecuación del equilibrio (ley de acción de masas) se deduce la ecuación de la curva de unión, curva de saturación o isoterma de unión:
| y, despejando B,: |
que es la ecuación de una hipérbola rectangular al representar B frente a F (unido frente a libre) (obsérvese la homología, por ej., con la ecuación de Michaelis-Menten).
La representación más asequible o directa es unido (variable medida) frente a total (variable establecida experimentalmente); su ecuación es más compleja, pero tiene una forma parecida.
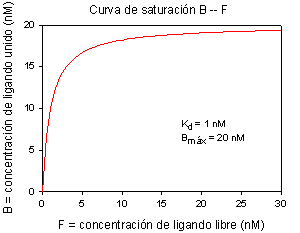 |
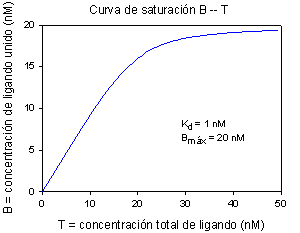 |
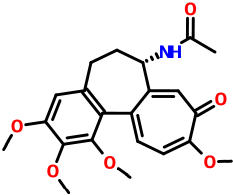
La colchicina, un alcaloide obtenido de la planta Colchicum autumnale (azafrán silvestre), se une específicamente a la proteína tubulina, principal componente de los microtúbulos.
Dicha unión evita la polimerización de la tubulina para formar microtúbulos; esto permite la aplicación experimental de la colchicina para estudiar procesos celulares dependientes de los microtúbulos (liberación de vasopresina por la médula renal, regulación de la secreción hepática, secreción por las células beta del páncreas, flujo axoplásmico...).
Un ensayo de unión podría diseñarse así:
| tubo nº | (en todos los tubos) | μL de agua | μL de [3H]colchicina |
|---|---|---|---|
| 0 |
150 μL de extracto de tejido
25 μL de GTP 1 mM
25 μL de NaCl 3.2 M |
50 | 0 |
| 1 | 49 | 1 | |
| 2 | 47 | 3 | |
| 3 | 44 | 6 | |
| 4 | 40 | 10 | |
| 5 | 35 | 15 | |
| 6 | 30 | 20 | |
| 7 | 20 | 30 | |
| 8 | 0 | 50 |
Tras añadir la colchicina en último lugar, se incuba 90 min a 35°C (tiempo suficiente, pues en ensayos previos se comprobó que el equilibrio de unión se alcanza en 35-40 min).
En todo ensayo de unión es esencial disponer de un método para separar el ligando unido del libre. En este caso, es la adsorción del ligando libre por el carbón activo.
Supongamos que se obtuvieron éstos:
| tubo | 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 |
|---|---|---|---|---|---|---|---|---|---|
| radiactividad (cpm) | 12 | 9 982 | 36 014 | 55 024 | 111 258 | 153 035 | 179 254 | 240 235 | 304 254 |
| tubo | 0’ | 1’ | 2’ | 3’ | 4’ | 5’ | 6’ | 7’ | 8’ |
| radiactividad (cpm) | 5 | 13 865 | 30 845 | 65 138 | 100 357 | 149 604 | 184 925 | 236 485 | 282 654 |
(donde 1 y 1’ son duplicados con la misma composición)
En el tratamiento que sigue, representaremos la radiactividad como R (RT = radiactividad total, RB = radiactividad unida).
1) Se calculan los valores de ligando total, unido y libre y se representan para obtener una curva de saturación:
Fórmulas para completar la tabla del ejercicio:
N = 0.2 μM × V = 0.2·10−6 mol/L × {V}·10−6 L = 0.2 × {V} x 10−12 mol = ( 0.2 × {V} ) pmol
las llaves quieren representar el valor numérico sin unidades, en este caso el de V
T = N / 250 μL = {N} pmol / 250·10−6 L = ({N} / 2.5 10−4 ) pM
RT = {T} 10−12 mol/L × 250·10−6 L × 60 Ci/10−3 mol × 2.22·1012 dpm/Ci × 0.55 cpm/dpm =
= ( {T} × 18.315 ) cpm
| {RB} cpm × 109 pmol/mol | ||
| B = | = ( {RB} / 18315 ) pM | |
| 250·10−6 L × 60 Ci/10−3 mol × 2.22·1012 dpm/Ci × 0.55 cpm/dpm |
F = T − B
2) Se representa B frente a F (opcional: se representa también B frente a T; en este caso es prácticamente lo mismo)
3) Hace falta ajustar a una curva los datos de B frente a F, para obtener los valores de Kd y Bmáx. El método clásico para hacer esto es transformar los datos para que la curva hiperbólica se convierta en una recta (representación de Scatchard) y ajustar ésta por mínimos cuadrados (regresión lineal). Hoy en día se pueden emplear programas de ordenador para ajustar directamente los datos a una hipérbola (regresión no lineal), lo cual es mucho más correcto desde el punto de vista matemático y estadístico (pues la transformación de los datos distorsiona su error experimental). De todos modos, la gráfica de Scatchard es útil para obtener información visual de la afinidad y la concentración de sitios de unión en el receptor, y para detectar la presencia de varios tipos de receptor o de una unión inespecífica (unión de baja afinidad no saturable).
Deducción teórica de la ecuación de Scatchard:
| Partimos de | |||
| y transformando: |
Es decir, la representación de B/F (unido / libre) frente a B (unido) (representación de Scatchard) proporciona una recta cuya pendiente negativa es igual a la constante de afinidad.
| Prolongando la recta hasta su corte con el eje de abscisas: | luego |
Es decir, la abscisa en el origen es igual a la concentración de sitios de unión.
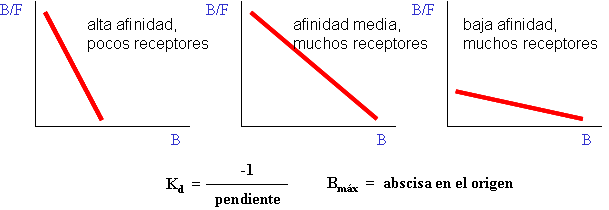
4) Se hacen los cálculos para representar la gráfica de Scatchard.
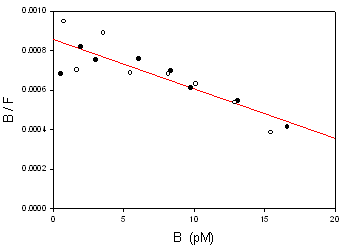
El ajuste por regresión lineal (mínimos cuadrados) proporciona:
pendiente = −Ka = −2.51·10−5 pM−1 ; Kd = 39840 pM
ordenada = 8.561·10−4
Bmáx = 8.561·10−4 / 2.51·10−5 = 34.11 pM
r² = 0.7612
NOTA: En este ejemplo, la fracción de ligando unida es muy baja (baja afinidad del receptor). Por ello, casi no hay diferencia entre F y T, ni entre sus gráficas respectivas. Además, debido a la escasa unión, no se consigue alcanzar la saturación, por lo que la estimación de Bmáx no es fiable (los datos experimentales no se aproximan al eje X en la gráfica Scatchard), es decir, el valor obtenido será muy impreciso.
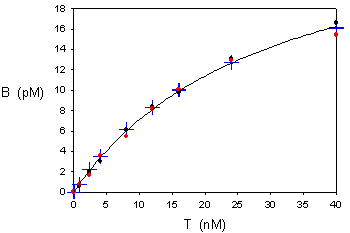
5) El ajuste por regresión no lineal proporciona estos resultados:
Bmáx = 28.34 ± 1.27 (error estándar), es decir, un 4.5% de error (C.V., coeficiente de variación)
Kd = 29745 ± 2344 (error estándar), es decir, un 7.9% de error
Los datos “experimentales” usados en este ejemplo se habían generado por simulación a partir de estos valores:
Bmáx = 27 pM, Kd = 27 nM = 27000 pM
Con lo que se aprecia que el segundo ajuste da valores más exactos.
Utilizan una menor cantidad de ligando marcado, que se mantiene fija, y cantidades crecientes de ligando frío. La curva de desplazamiento, B frente a [L´], es sigmoidal y decreciente.
El ligando frío es el mismo que el radioligando. Ofrecen utilidad similar a las curvas de saturación.
El ligando frío es diferente al marcado. Se usan en especial para estudiar la potencia de diversos análogos o inhibidores, que se calcula a partir del punto central de la curva, en forma de “dosis eficaz 50%” o “concentración inhibidora 50%” (IC50). Este valor está relacionado con la constante de equilibrio de unión del competidor (KI) al receptor.