
 )
está formado por 8 cadenas polipeptídicas, con una estructura
(γ2)(αβ)(αβ)(γ2)
)
está formado por 8 cadenas polipeptídicas, con una estructura
(γ2)(αβ)(αβ)(γ2)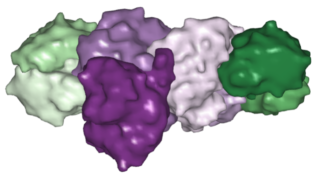
Este complejo enzimático
(EC 1.18.6.1
 )
está formado por 8 cadenas polipeptídicas, con una estructura
(γ2)(αβ)(αβ)(γ2)
)
está formado por 8 cadenas polipeptídicas, con una estructura
(γ2)(αβ)(αβ)(γ2)
 En tonos verdes las subunidades γ de la reductasa y en violetas las αβ de la dinitrogenasa.
En tonos verdes las subunidades γ de la reductasa y en violetas las αβ de la dinitrogenasa.
Esquemáticamente:
Analicemos sus cofactores y el mecanismo de su acción catalítica:


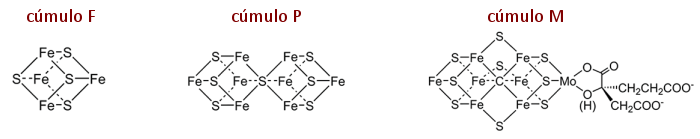
 con un cúmulo Fe7MoS9 (FeMoco, cofactor FeMo, o cúmulo M),
con un cúmulo Fe7MoS9 (FeMoco, cofactor FeMo, o cúmulo M), coordinado por un residuo de cisteína, otro de histidina y una molécula de homocitrato
coordinado por un residuo de cisteína, otro de histidina y una molécula de homocitrato  con un cúmulo Fe8S7 (o cúmulo P),
con un cúmulo Fe8S7 (o cúmulo P),  coordinado por 6 residuos de cisteína
coordinado por 6 residuos de cisteína
 con un cúmulo Fe4S4 en común (o cúmulo F),
con un cúmulo Fe4S4 en común (o cúmulo F),  coordinado por 4 residuos de cisteína
coordinado por 4 residuos de cisteína
Los electrones (probablemente originados desde NADH o NADPH) se transfieren, de uno en uno, desde la ferredoxina a la reductasa y de ésta a la dinitrogenasa. Cuando el cofactor FeMo ha captado 2 e− oxida 2 H+ generando H2; ello permite la unión del N2. Tras recibir el cofactor FeMo otros 6 e− se produce la reducción del N2 hasta 2 moléculas de NH3:
![]() Estructura molecular 3D del complejo nitrogenasa completo (ampliación)
Estructura molecular 3D del complejo nitrogenasa completo (ampliación)
Estructuras extraídas de 1M34.pdb, doi:10.2210/pdb1m34/pdb, complejo nitrogenasa de Azotobacter vinelandii estabilizado con ADP-tetrafluoroaluminato. Schmid et al. 2002, difracción de rayos X.
[ Fe4S4 ] InChI Key: LJBDFODJNLIPKO-VKOJMFJBAC
[ Fe7MoS9 ] InChI Key: UZRXIPMKRKMLQF-VGXFRCDIAP
[ Fe8S7 ] InChI Key: JKVMXLBGZBULKV-DHHOTQGYAO
Bibliografía: doi:10.1146/annurev.biochem.78.070907.103812 (2009), doi:10.1021/cr400641x (2014)