- http://www.geneq.com/catalog/en/sba.html
Applications example:
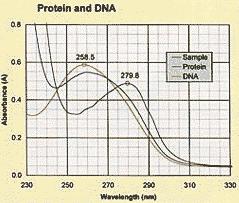
Espectros experimentales de proteína y DNA.
El dato de partida fue el archivo de imagen de:

Manualmente se generaron curvas para que ajustasen bien visualmente con las dos curvas experimentales. Se empleó para ello SigmaPlot usando User-Defined-Transforms en las que se combinaron ecuaciones copiadas de la biblioteca de SigmaPlot (Graph > Plot Equation > Library > Peak )
En SigmaPlot se generaron datos (X,Y) para cada 2 nm. Combinando 1 lorentziana (para la línea de fondo) y 2 gaussianas se superpone razonablemente bien con los espectros experimentales.
Proteínas:
y = 0.032 + 0.395 * exp(-0.5*((x-281)/11)^2)
+ 0.125 * exp(-0.5*((x-262)/9)^2)
+ 3.5 / (1+((x-228)/6.2)^2)
DNA:
y = 0.066 + 0.480 * exp(-0.5*((x-258)/13.5)^2)
+ 0.158 * exp(-0.5*((x-279)/10)^2)
+ 6.2 / (1+((x-200)/6.2)^2)
El DNA daba A260/A280 = 1.63; lo cambié a y = 0 + 0.505 * etc. para conseguir un cociente de 1.8,
y el de proteínas también a y = 0 + etc.
Con ellos puedo crear una función generadora de espectros combinados.
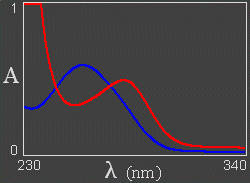 Siguiendo un desarrollo parecido al utilizado con los espectros de dicroísmo circular, se empleó la biblioteca gráfica en JavaScript de Walter Zorn para conseguir el dibujo de la gráfica.
Siguiendo un desarrollo parecido al utilizado con los espectros de dicroísmo circular, se empleó la biblioteca gráfica en JavaScript de Walter Zorn para conseguir el dibujo de la gráfica.
Los datos empleados para construir las gráficas son:
Angel Herráez. Parte de la sede web Biomodel.uah.es