
 Curso de Posgrado:
Curso de Posgrado:
Análisis in vitro e in silico de ADN y proteínas
Universidad Nacional de La Pampa, 1 a 4 y 9 a 11 de diciembre de 2015

Profesor: Dr. Angel Herráez - Universidad de Alcalá (España)
El curso, de carácter teórico y práctico, ocupa 7 días con un total de 60 horas lectivas y se desarrolló en la Sala III de Computación, Facultad de Ciencias Exactas y Naturales (Santa Rosa).
Universidad Nacional de La Pampa, Programa Estímulo de Formación de Posgrados, Subprograma II: Dictado de cursos o seminarios de posgrado a cargo de especialistas del exterior. Convocatoria 2015. (Resolución nº 191/15, del Consejo Superior UNLPam.
Aprobado por Resolución nº 311/15 del Consejo Superior UNLPam y Resolución nº 396/15 del Consejo Directivo de la Facultad de Ciencias Exactas y Naturales.
Fue impartido por Angel Herráez, profesor titular de Bioquímica y Biología Molecular en la Universidad de Alcalá.
El curso es gratuito para docentes y graduados de la UNLPam.
Algunos comentarios de los asistentes tras el curso:
- Es mi primer curso de postgrado ¡espero que todos los siguientes sean así de interesantes! ¡Me queda un lindo recuerdo!
- El curso fue muy interesante. Y sé que me a a servir para seguir con el estudio de la proteína en cuestión.
- El curso me gustó mucho, el tema muy interesante y muy útil para muchas aplicaciones de la Química, Biología, investigación y docencia, y la forma de explicar los temas excelentes.
- Le quiero agradecer mucho su amabilidad y respeto para con nosotros
durante el transcurso de las clases, la verdad aprendimos mucho.
Contenidos:
- Introducción
- Cómo estudiar el DNA.
2a) Fundamento de las técnicas de secuenciación de DNA.
2b) Detección de la mutación causante de la anemia drepanocítica mediante secuenciación del DNA. (Ejercicio práctico in silico)
2c) Ensayos de hibridación de ácidos nucleicos: Southern, northern, FISH.
2d) Fundamento, variantes y aplicaciones de las técnicas de PCR y PCR cuantitativa.
2e) Análisis de polimorfismos genéticos.
2f) Diagnóstico genético del genotipo responsable de la anemia drepanocítica. (Ejercicio práctico in silico)
2g) Ensayo de paternidad empleando marcadores polimórficos STR CODIS, mediante PCR múltiplex y análisis electroforético. (Ejercicio práctico in silico)
2h) Micromatrices, microarreglos o chips de DNA.
- Análisis de la interacción entre proteínas y DNA.
3a) Mecanismos moleculares de la unión de proteínas al DNA y la especificidad por secuencias diana.
3b) Motivos estructurales proteicos implicados en la unión al DNA: HTH, HLH, homeodomino, dedos de zinc, cremallera de leucina. Análisis tridimensional de su estructura. (Estudio in silico)
3c) Acción de los factores de transcripción en la regulación de la expresión génica.
3d) Técnicas para la localización de secuencias diana de una proteína y para la detección de proteínas ligantes de DNA: southwestern, northwestern, retardo en gel (EMSA), huella con DNasa (DNA footprinting), análisis de series de deleción, rastreo con mutaciones.
3e) Interpretación de experimentos de localización de dianas y de detección de proteínas ligantes de DNA. (Ejercicio)
- Qué información contienen las bases de datos bioinformáticas y cómo aprovecharla
4a) Búsqueda de secuencias de ácidos nucleicos y proteínas: exploración de las bases de datos NCBI GQuery:Nucleotide, Gene y otras.
4b) Búsqueda y análisis de la estructura molecular de ácidos nucleicos y proteínas: exploración de las bases de datos PDB y NDB.
4c) Búsqueda de metabolitos, ligandos, fármacos y otros agentes químicos en la base de datos PubChem.
- Cómo visualizar la estructura tridimensional de las moléculas y análisis de estructuras moleculares
5a) Aplicaciones como parte del proceso de investigación y como herramienta docente y de presentación.
5b) Aprendizaje del software: principalmente Jmol y JSmol.
5c) Presentación y uso de portales: PDB-101 y Proteopedia.
5d) Investigación de las características funcionales de las moléculas mediante análisis de su estructura 3D.
- Cómo mostrar las estructuras moleculares con una perspectiva tridimensional - Taller de aprendizaje práctico.de estructuras moleculares con una perspectiva tridimensional
6a) Uso personalizado de las plataformas existentes, tanto para acceder a su información como para autoría y presentación.
6b) Obtención de estructuras moleculares en las bases de datos públicas.
6c )Manejo del software Jmol/JSmol para mostrar e interaccionar con las estructuras.
6d) Autoría y presentación de estructuras moleculares: Representación personalizada de modelos moleculares. Presentación dentro de Proteopedia.
6e) Preparación de modelos para mostrarlos usando "realidad aumentada".
6f) Presentación al grupo de los miniproyectos personales elaborados durante el curso.
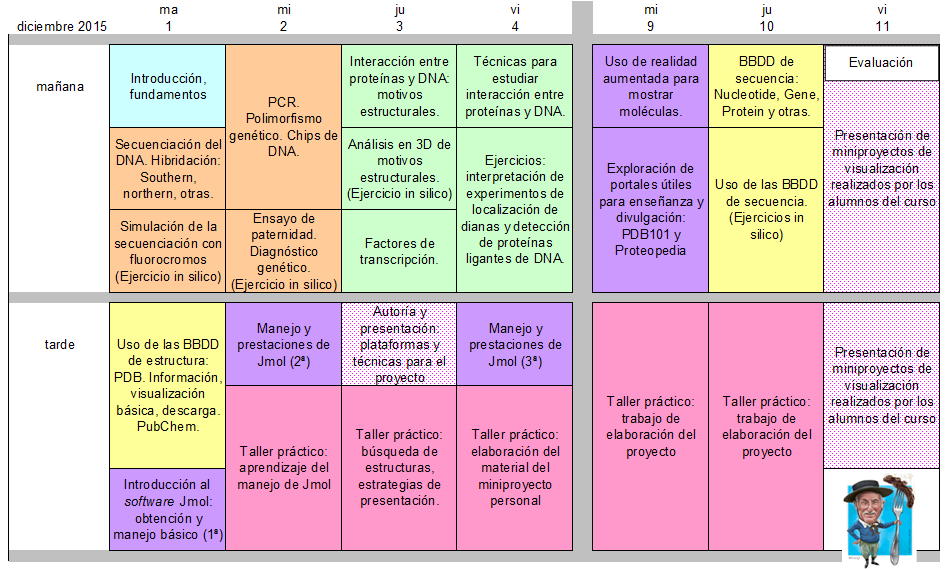
Cronograma:

 Curso de Posgrado:
Curso de Posgrado:

 Curso de Posgrado:
Curso de Posgrado:

