15.4 Fundamento y ejemplos del uso de genes marcadores para la detección y selección de clones
Con el término genes marcadores (pág. 232) se hace referencia a segmentos de DNA que se incorporan al DNA recombinante para facilitar las tareas de
- detección de aquellos clones celulares que han incorporado el vector o, preferiblemente, el DNA recombinante deseado,
- selección de los mismos clones, es decir, conseguir que sobrevivan y proliferen las células que han incluido el recombinante mientras que las que no lo hayan hecho mueran o, al menos, no proliferen.
Aunque el gen marcador puede incorporarse en cada caso como un inserto más (análogo al inserto auxiliar para clonación con expresión, pág. 220), habitualmente el gen marcador forma ya parte del vector elegido (vectores comerciales optimizados para la clonación, preparados a su vez por ingeniería genética, como el plásmido de pág. 226).
El producto codificado por el gen marcador manifiesta un fenotipo que se puede detectar o bien es responsable de la supervivencia o muerte de la célula, o de su proliferación.
Detección: ensayo de colonias blancas y azules
Un gen marcador de uso frecuente es lacZ, el gen de la β-galactosidasa. Esta enzima hidroliza la lactosa en galactosa + glucosa, pero en el laboratorio se aprovecha su capacidad para hidrolizar también el sustrato sintético conocido como X-gal; la actividad enzimática se detecta fácilmente por el color azul del producto de la reacción.

|
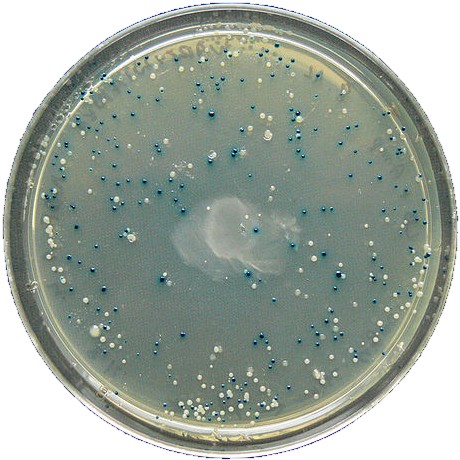
Si el DNA recombinante incluye el gen lacZ, la célula anfitriona producirá galactosidasa. Cuando se añade X-gal al medio de cultivo, se observa una coloración azul en los clones celulares ("colonias azules"). Las células que no hayan incorporado el recombinante no se teñirán de azul ("colonias blancas").
Este gen marcador no permite, obviamente, la selección (pues las células sobreviven en cualquier caso) sino tan solo la detección. La selección deberá hacerse manualmente, extrayendo las colonias adecuadas y transfiriéndolas a una nueva placa o recipiente donde se continúa su cultivo.
Selección: genes de resistencia
Se trata de uno de los tipos de genes marcadores que se utilizan con más frecuencia para la selección de clones recombinantes, particularmente en bacterias. La transcripción y traducción del gen en la célula anfitriona conduce a una proteína que permite a la célula sobrevivir en presencia de un antibiótico.
Gen de resistencia a ampicilina (ampr)
La ampicilina es un antibiótico de la familia beta-lactamas (como la penicilina) que actúa sobre diversas bacterias impidiendo que sinteticen el peptidoglicano de la pared celular. Sin dicha pared, las bacterias no completan su división, pues se lisan y mueren.
El gen ampr codifica una β-lactamasa (E.C.3.5.2.6) que degrada la ampicilina. Cuando se incuban las bacterias en un medio con ampicilina, sólo aquéllas que hayan incorporado el gen marcador como parte del DNA recombinante pueden, por ello, sobrevivir y proliferar.
Gen de resistencia a tetraciclina (tetr, tetA)
La tetraciclina se une a la subunidad menor (30S) del ribosoma bacteriano, impidiendo la entrada de los aminoacil-tRNA y, así, la síntesis de proteínas. Las bacterias no pueden proliferar sin sintetizar sus proteínas. En condiciones normales la tetraciclina se concentra dentro de la bacteria gracias a un transporte activo.
El gen tetA codifica una proteína que se asocia a la membrana de la bacteria y bombea moléculas de tetraciclina hacia el exterior (mecanismo de eflujo). Cuando se incuban las bacterias en un medio con tetraciclina, sólo aquéllas que hayan incorporado el gen marcador como parte del DNA recombinante pueden, por ello, sobrevivir y proliferar.
Gen de resistencia a neomicina (neo)
La neomicina es un antibiótico de la familia estructural de los aminoglicósidos (a la que pertenece, entre otros, la estreptomicina). Interfiere en la iniciación y la elongación de la traducción.
Los genes neo codifican una aminoglicósido-3'-fosfotransferasa (EC 2.7.1.95), que fosforila el aminoglicósido a costa de ATP. El antibiótico fosforilado no es captado por las células, lo que neutraliza su acción.
Las bacterias se incuban en un medio con neomicina o kanamicina (otro aminoglicósido), de modo que sólo aquéllas que hayan incorporado el gen marcador como parte del DNA recombinante sobrevivan y proliferen.
Este marcador se usa asimismo con células eucarióticas, en cuyo caso la selección se consigue habitualmente con el antibióitico G148 (Geneticin®), que afecta a la traducción eucariótica.
Selección: complemento metabólico
En este caso, se emplean células anfitrionas que poseen una mutación que les imposibilita el crecimiento en un determinado medio de cultivo. El defecto se suple gracias a un gen incorporado en el recombinante. Esta aproximación es frecuente cuando se han de emplear células anfitrionas eucarióticas.
Síntesis de triptófano: fosforribosil-antranilato isomerasa
Las levaduras de una línea celular mutante carente de esta enzima, que cataliza una de las etapas en la ruta de biosíntesis de triptófano, sólo sobreviven si disponen de triptófano. Cultivando en un medio carente de este aminoácido sólo proliferarán las células que hayan incorporado el DNA recombinante que aporta el gen TRP1 que codifica dicha enzima.
Síntesis de nucleótidos: dihidrofolato reductasa
Las células mutantes sin actividad dihidrofolato reductasa (un fenotipo que se indica como dhfr¯) requieren un suministro de timidina, glicina y purinas para poder sintetizar sus nucleótidos y proliferar. Si no se incluyen estos 3 precursores en el medio de cultivo los clones sólo sobreviven cuando han incorporado un DNA recombinante que contenga el gen dhfr.
Es también posible usar células normales, no mutadas, añadiendo al medio metotrexato, un análogo estructural del tetrahidrofolato que inhibe la dihidrofolato reductasa. Las células no transformadas no sobreviven (a falta de timidina, glicina y purinas), pero si habían captado el gen dhfr como parte de un DNA recombinante pueden compensar la inhibición mediante una mayor prroducción de la enzima y sobrevivir (se dice que son resistentes al metotrexato).
Síntesis de nucleótidos: timidina quinasa
Las células mutantes sin actividad timidina quinasa (tk¯) no sobrevien en un medio HAT (con hipoxantina, aminopterina y timidina). Este medio bloquea la "vía de recuperación" para la síntesis de nucleótidos, mientras que la timidina quinasa es esencial para la "vía de novo". En consecuencia, sólo los clones que hayan incorporado un DNA recombinante que contenga el gen tk funcional podrán sobrevivir en este medio.
Puesto que es difícil conseguir incorporar en un mismo vector un inserto (gen o fragmento) a clonar y un gen marcador de selección, junto con las respectivas regiones promotoras eucarióticas —necesarias para que se expresen—, lo más frecuente es acudir a la cotransformación (pág. 230) de las células con dos DNA recombinantes preparados por separado, uno que incluye el gen marcador en un vector y el otro con el DNA de interés incorporado en otra molécula de vector.
Detección: inactivación insercional
Los genes marcadores como lacZ son útiles pero presentan el problema de que, aunque permiten detectar las células que han incorporado el DNA, en realidad sólo indican que se ha incorporado el vector, sea éste recombinante o no —es decir, tanto si tiene el inserto incluido como si es un vector "vacío".
Referencias:
- Imagen de la placa con colonias bacterianas: Wikimedia Commons [1] - licencia cc-by-sa; autor: Navaho.