LDH: lactato deshidrogenasa (deshidrogenasa del l-lactato); EC 1.1.1.27 ; (S)-lactato:NAD oxidorreductasa
Cataliza reversiblemente la reacción (S)-lactato + NAD  piruvato + NADH + H+
piruvato + NADH + H+
Existen dos genes que codifican subunidades componentes de la LDH. En el ser humano se llaman gen LDHA y gen LDHB.
Gen LDHASituado en el cromosoma 11, posición 11p15.1 |
Gen LDHBSituado en el cromosoma 12, posición 12p12.1 |
transcripción + traducción |
transcripción + traducción |
cadena polipeptídica "A" o "M"  |
cadena polipeptídica "B" o "H"  |
Ambos polipéptidos, "A" o "M" y "B" o "H", son muy parecidos en su estructura, pero tienen algunas diferencias de secuencia:
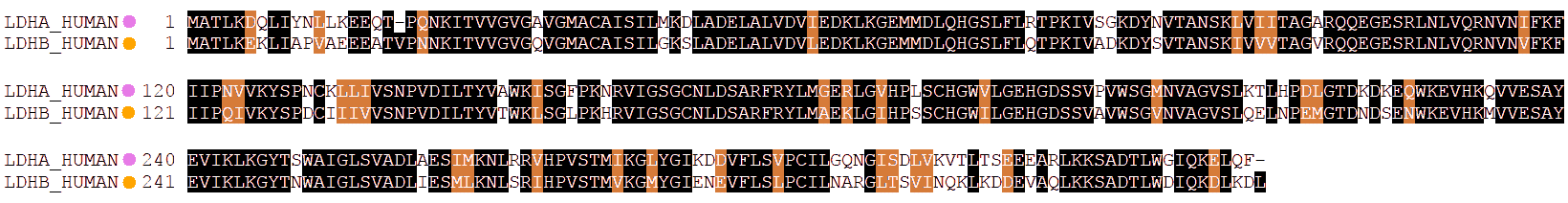
Como consecuencia, sus propiedades fisicoquímicas son un poco diferentes; por ejemplo, su punto isoeléctrico y sus propiedades cinéticas como enzimas.
La enzima LDH activa es un tetrámero: está formada por 4 subunidades que pueden ser del tipo M o del H. Pueden, por tanto, asociarse en 5 combinaciones diferentes:
M4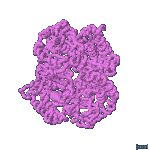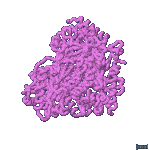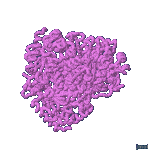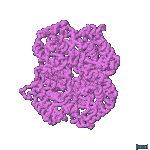 |
M3H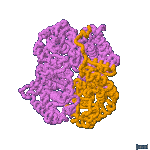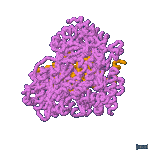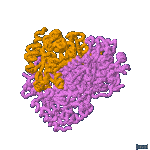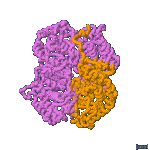 |
M2H2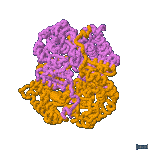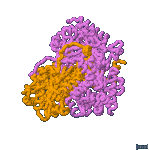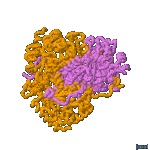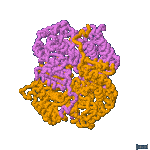 |
MH3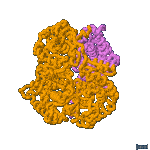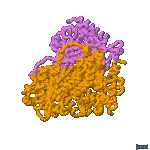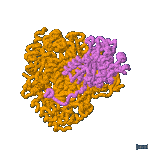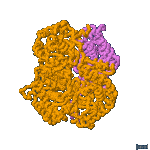 |
H4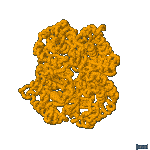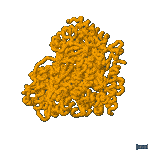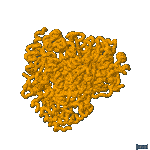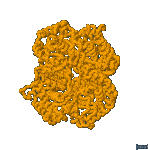 |
que constituyen las 5 isoenzimas de LDH.
Dependiendo de que en una célula se exprese en mayor o menor medida cada uno de los dos genes, se sintetizarán distintas cantidades relativas de los polipéptidos A y B, y la abundancia relativa de las 5 isoenzimas será diferente.
Ensaya cambios en la expresión relatva de los genes LDHA y LDHB (moviendo el control deslizante) y observa el resultado:
|
mayor expresión de LDHA
mayor expresión de LDHB
Cantidades de cada isoenzima:
M4
M3H
M2H2
MH3
H4
|
Ejemplos: En el y en el
se expresa predominantemente el gen LDHA, que codifica la subunidad M.
|
Angel Herráez. Parte de la sede web Biomodel.uah.es
Las imágenes animadas se han producido empleando Jmol en Proteopedia (2FM3.pdb)
Las secuencias se obtuvieron en UniProtKB; se calculó el alineamiento en T-Coffee y se representó usando BoxShade (ExPASy).