Attention:
La question 17 (série de questions B) pose un problème auquel on ne peut répondre que si le tutoriel utilise l'un des deux modèles d'ADN disponibles (voir l'option “a” ci-dessous). Le gestionnaire de site ou l'enseignant peuvent décider du modèle moléculaire à utiliser en en procédant simplement à l'édition de l'un des fichiers (ajout ou retrait de //). Voir l'explication détaillée ci-dessous.
Les réponses aux questions sont disponibles pour les instructeurs de ce lien.
(Approfondissement)
Deux modèles d'ADN sont disponibles au choix pour ce tutoriel. Vous pouvez facilement savoir lequel est en cours d'utilisation en ouvrant le pop-up menu de Jmol (clic droit dans la fenêtre, ou clic sur le logo Jmol dans le coin inférieur droit de la fenêtre Java) et en consultant le premier item du menu déroulant, la molécule indiquée peut être soit 1D66 soit dna.
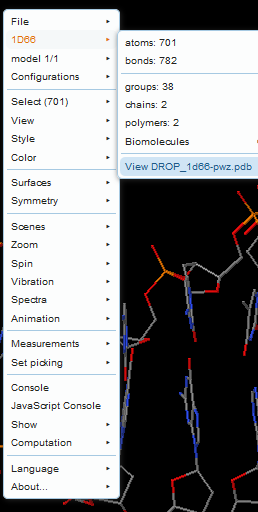 (a) Le classique 1d66-pwz.pdb (modifié par Eric Martz à partir de 1d66.pdb, pour l'ancien tutoriel ADN utilisant Chime).
(a) Le classique 1d66-pwz.pdb (modifié par Eric Martz à partir de 1d66.pdb, pour l'ancien tutoriel ADN utilisant Chime).
Ce modèle comporte une erreur en C28, dont la base est inversée de telle sorte que c'est le mauvais côtéqui se trouve face à la base complémentaire G11.
En utilisant les capacités de Jmol, de longues liaisons hydrogène sont tracées jusqu'aux bons atomes, C28 et G11, qui sont assez éloignés dans le modèle ; les liaisons hydrogène en résultant, anormalement longues, sont similaires à celles que dessine automatiquement Chime.
Martz a profité de cette anomalie pour proposer l'une de ses questions aux étudiants (série B, question n° 17)
Pour utiliser ce modèle moléculaire : ouvrez le fichier utils.js en utilisant un éditeur de texte et, près de la ligne 29, effacez les deux slashes (//) avant var DNAmodel = "1d66-pwz.pdb" et ajoutez-en deux avant var DNAmodel = "dna.pdb". Sauvegardez les modifications et rechargez la page du tutoriel.
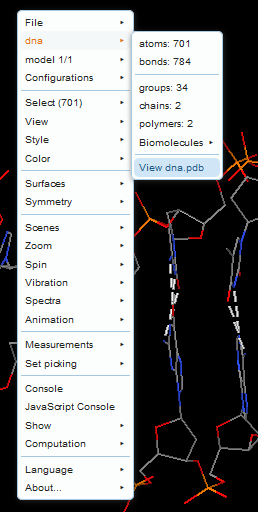 (b) Un nouveau dna.pdb (modifié par Angel Herráez à partir de 1d66-pwz.pdb) avec une correction de la position de C28, qui établit désormais des liaisons hydrogène normales (selon Watson-Crick) avec Jmol.
(b) Un nouveau dna.pdb (modifié par Angel Herráez à partir de 1d66-pwz.pdb) avec une correction de la position de C28, qui établit désormais des liaisons hydrogène normales (selon Watson-Crick) avec Jmol.
La question n° 17 n'a plus aucun sens avec cette molécule et il n'est plus possible d'y répondre.
Pour utiliser ce modèle moléculaire : ouvrez le fichier utils.js en utilisant un éditeur de texte et, près de la ligne 29, ajoutez deux slashes (//) avant var DNAmodel = "1d66-pwz.pdb" et effacez les deux slashes avant var DNAmodel = "dna.pdb". Sauvegardez les modifications et rechargez la page du tutoriel.