Achtung:
Frage 17 (in Fragenkomplex B) ist nur dann sinnvoll und beantwortbar, wenn das Tutorial ein bestimmtes von zwei angebotenen DNA-Modellen benutzt. Der Webautor oder Lehrer kann durch eine einfache änderung in einer der Dateien selbst entscheiden, welches der beiden Modelle benutzt wird. Ausführliche Erklärung.
Antworten zu den Fragen sind für Lehrpersonal bei diesem Link.
(fortgeschritten)
Zwei alternative DNA-Modelle werden für dieses Tutorial angeboten. Sie können prüfen, welches Modell gerade verwendet wird, indem Sie das Pop-up-Menü in Jmol öffnen (Klick mit rechter Maustaste, oder Klick auf das Jmol-Logo in der unteren rechten Ecke des Applets). Der oberste Eintrag lautet entweder 1D66 oder dna.
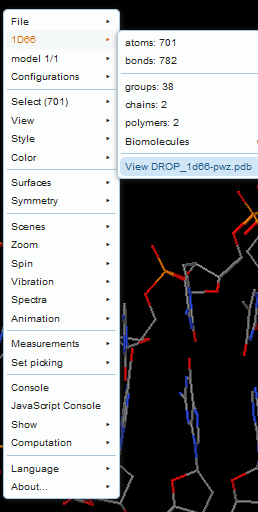 (a) Das klassische 1d66-pwz.pdb (modifiziert durch Eric Martz auf der Grundlage von 1d66.pdb, welches für das alte DNA-Tutorial unter Benutzung von Chime verwendet wurde).
(a) Das klassische 1d66-pwz.pdb (modifiziert durch Eric Martz auf der Grundlage von 1d66.pdb, welches für das alte DNA-Tutorial unter Benutzung von Chime verwendet wurde).
Dieses Modell hat einen Fehler in C28, dessen Base verdreht ist, so dass die falsche Seite dem komplementären G11 gegenüber liegt.
Mit Hilfe der Fähigkeiten von Jmol werden lange H-Bindungen zu den korrekten Atomen in C28 and G11 gezogen, die im Modell weit auseinander liegen; die resultierenden, anomal langen H-Bindungen sind gleich jenen, die automatisch durch Chime gezogen werden.
Martz nutzte diese Anomalie zur Formulierung einer der Fragen für Studenten (Nr. 17).
Zur Benutzung dieses Modells öffnen Sie die Datei utils.js mit einem Text-Editor; entfernen Sie in (oder in der Nähe von) Zeile 29 die zwei Schrägstriche (//) vor var DNAmodel = "1d66-pwz.pdb" und fügen Sie zwei neue Schrägstriche vor var DNAmodel = "dna.pdb" ein. Speichern Sie die änderungen und öffnen Sie erneut die Tutorial-Seite.
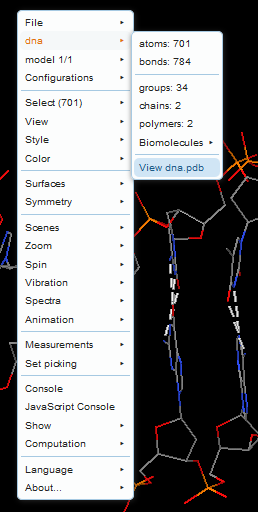 (b) Ein neues dna.pdb (modifiziert durch Angel Herráez auf der Grundlage von 1d66-pwz.pdb) mit korrigierter Position von C28, das nun normale Watson-Crick-H-Bindungen in Jmol zeigt.
(b) Ein neues dna.pdb (modifiziert durch Angel Herráez auf der Grundlage von 1d66-pwz.pdb) mit korrigierter Position von C28, das nun normale Watson-Crick-H-Bindungen in Jmol zeigt.
Frage 17 hat keinen Sinn für dieses Modell und kann nicht beantwortet werden.
Zur Benutzung dieses Modells öffnen Sie die Datei utils.js mit einem Text-Editor; fügen Sie in (oder in der Nähe von) Zeile 29 zwei Schrägstriche (//) vor var DNAmodel = "1d66-pwz.pdb" hinzu und entfernen Sie die zwei Schrägstriche vor var DNAmodel = "dna.pdb". Speichern Sie die änderungen und öffnen Sie erneut die Tutorial-Seite.